سبيل ترميز JAK-STAT هو سلسلة من التفاعلات بين البروتينات في خلية، ويتخل في عمليات مختلفة مثل المناعة وانقسام الخلية وموت الخلية وتشكل الورم. ينقل السبيل معلومات من إشارات كيميائية خارج الخلية إلى نواة الخلية مسبباً تفعيل مورثات عبر عملية تدعى الانتساخ. هناك ثلاثة أجزاء رئيسة من ترميز JAK-STAT: أنزيمات Janus kinases(JAKs) ومحول إشارة مفعِّل لبروتينات الانتساخ (STATs) ومستقبلات(تربط الإشارات الكيميائي). [1] قد يؤدي اضطراب سبيل JAK-STAT إلى أمراض عديدة مثل الأمراض الجلدية والسرطانات والاضطرابات المؤثرة على جهاز المناعة.
بنية JAKs و STATs
المقالات الرئيسية : JAKs و STATs
هناك 4 بروتينات JAK: JAK1 و JAK2 و JAK3 و TYK2 . [1] تحتوي JAKs على نطاق FERM (حوالي 400 وحدة) ، و نطاق مرتبط بـSH2 (مئة وحدة تقريباً) ونطاق كيناز (مئتان وخمسون وحدة تقريباً) ونطاق بسودوكيناز(ثلاثمئة وحدة تقريباً). [2] نطاق الكيناز أساسي لنشاط JAK لأنه يسمح للـ JAKs بفسفرة(إضافة مجموعات الفوسفات) البروتينات.
هناك 7 البروتينات STAT: STAT1 ، STAT2 ، STAT3 ، STAT4 ، STAT5A ، STAT5B و STAT6 . [1] تحتوي بروتينات STAT على الكثير من النطاقات المختلفة لكل منها وظيفة مختلفة وأكثر المواقع ثباتاً(انحفاظاً) فيها هو نطاق SH2 . [2] يتكون نطاق SH2 من حلزوني ألفا وصحيفة بيتا وتتشكل تقريباً من وحدات 575-680. [3] لبروتينات STATs أيضاً نطاقات تنشيط انتساخية(TAD)، وهي أقل انحفاظاً وتتوضع على النهايةC. [4] علاوة على ذلك، تحتوي الـSTATs أيضاً على تفعيل التيروزين ونهايات أمينية ووصلات ملتفة ونطاقات رابطة للدنا.
الآلية

يتسبب ارتباط جزيئات مختلفة- سيتوكينات عادة- مثل الإنتفرونات والإنترلوكينات بمستقبلات سطح الخلية بتحول المستقبلات إلى مثنويات، ما يستحضر الـJAKs المرتبطة بالمستقبل إلى مسافة قريبة جداً. [5] تفسفر الـ JAKs بعد ذلك بعضها على جزيئات تيروزين تتوضع في مناطق تدعى عُرى التنشيط عبر عملية تدعى الفسفرة الفسفرة الانتقالية (transphosphorylation) ما يزيد نشاط سلاسل الكيناز خاصتها. تفسفر الـJAKs المنشطة بعد ذلك وحدات التيروزين على المستقبل صانعة مواقع للبروتينات الحاوية على سلاسل SH2. ترتبط الـSTATs بعد ذلك بالتيروزينات المفسفرة على المستقبل باستخدام سلاسل SH2خاصتها ثم يفسفر التيروزين خاصتها بواسطة JAKs ما يسبب انفصال الـ STATs عن المستقبل. [2] تشكّل هذه الـSTATs المنشطة مثنويات أحادية أو عديدة حيث يربط نطاق SH2 من كل STAT التيروزين المفسفر من STAT المقابل، لينتقل المثنوي بعدها إلى نواة الخلية ليحرض انتساخ الجينات المستهدفة. قد يتفسفر تيروزين الـSTATs مباشرة بكيناز تيروزين المستقبل، لكن وبسبب افتقار معظم المستقبلات للكيناز البنيوي، عادة ما تكون هناك حاجة لـ JAKs من أجل الترميز. [1]
انتقال STATs من العصارة الخلوية إلى النواة
يجب على مثنويات STAT من أجل الانتقال من العصارة الخلوية إلى النواة أن تعبر عبر المعقدات المسامية النووية (NPCs) وهي معقدات بروتينية على طول المغلف النووي تتحكم بتدفق المواد الداخلة والخارجة من وإلى النواة. لتمكين STATs من الدخول إلى النواة، ترتبط سلسلة أمينية على STATs-تُدعى إشارة التحديد النووية (NLS) ببروتينات تُدعى الإمبورتين. [4] حالما تدخل مثنويات STAT (المرتبطة بالإمبورتين) إلى النواة، يرتبط بروتين يُدعى RAN (مرتبط بـ GTP) بالإمبورتين ليحررها من مثنوي STAT. [6] يصبح مثنوي الـSTAT حراً في النواة بعدها.
يبدو أن بعض الـ STATs بالتحديد ترتبط ببعض الإمبوتين بالتحديد. مثلاً تستطيع بروتينات STAT3 الدخول إلى النواة بالارتباط بـ importin α 3 وimportin α6. [7] بينما ترتبط STAT1 وSTAT2 بـ importin α5. [4] تظهر الدراسات أن STAT2 يحتاج إلى بروتين يُدعى عامل تنظيم إنترفيرون 9 IRF9 ليدخل إلى النواة. [6] لا يُعرف الكثير عن دخول STATs الأخرى إلى النواة لكن من المُفترض أن سلسلة من الحموض الأمينية على السلسلة الرابطة للدنا في STAT4 قد تسمح بالنقل النووي. كما يمكن لكل من STAT5 وSTAT6 أن يرتبطا بالإمبورتين α3. بالإضافة لذلك يستطيع STAT3 وSTAT5 وSTAT6 الدخول إلى النواة حتى بدون فسفرة نهايات التيروزين.
دور تعديلات ما بعد الانتقالية
بعد صنع STATs بتركيب البروتينات الحيوي ترتبط بها جزيئات غير بروتينية تُدعى بالتعديلات بعد الانتقالية. أحد هذه الأمثلة هو فسفرة التيروزين (وهي أساسية لترميز JAK-STAT) لكن تتعرض STATs لتعديلات أخرى قد تؤثر على سلوكها في سبيل ترميز JAK-STAT. تتضمن هذه التعديلات المثيلة والأستلة وفسفرة السيرين.
- المثيلة. يمكن أن يتعرض STAT3 لمثيلة ثنائية (مجموعتي ميثيل) على نهاية الليسين. في الموقع 140 ويُعتقد أن ذلك يخفض نشاط STAT3. [8] هناك جدال بشأن مثيلة STAT1 على نهاية الأرجنين (في الموقع 31) وما الوظيفة الممكنة لهذه المثيلة. [9]
- الأستلة. أظهرت STAT1 وSTAT2 وSTAT3 وSTAT5 وSTAT6 تعرضاً للمثيلة. [10] ربما ترتبط مجموعة أستيل بالليزين على STAT1 في الموقع 410 و413 لتحرض بالنتيجة انتساخ مورثات الموت الخلوي المبرمج محرضة على موت الخلية. أستلة STAT2 مهمة للتفاعلات مع STATs أخرى ولانتساخ المورثات المضادة للفيروسات. [4]
اُقترح أهمية أستلة STAT3 ليصبح مثنوياً وللارتباط بالدنا والقدرة على ترجمة المورثات، كما تتطلب سبل IL-6 JAK-STAT التي تستخدم STAT3 الأستلة لانتساخ مورثات الاستجابة لـ IL-6. [10] أستلة STAT5 على الليزين في الموقعين 694 و701 من أجل تحويل STAT إلى مثنويات في ترميز البرولاكتين. [11] يُعتقد أن إضافة مجموعة أستيل إلى STAT6 أساسية من أجل انتساخ المورثات في بعض أشكال ترميز IL-4 لكن لا تُعرف كل الحموض الأمينية المؤستلة على STAT6.
- فسفرة سيرين - تخضع أغلب STATs السبع (باستثناء STAT2) لفسفرة السيرين. [2] أظهرت فسفرة STATs تقليل انتساخ المورثات. [12] كما أنها ضرورية من أجل انتساخ بعض المورثات الهدفية للسيتوكينات IL-6 وIFN-γ . [9] اُفترض تأثير فسفرة السيرين على تنظيم تثاني STAT1، وأن فسفرة السيرين المستمرة في STAT3 تؤثر على انقسام الخلية. [13]
توظيف المنشطات المساعدة
ككثير من عوامل الانتساخ الأخرى، تستطيع STATs توظيف منشطات مساعدة مثل CBP وp300 وتزيد هذه المنشطات المساعدة من انتساخ المورثات المستهدفة. [2] تستطيع المنشطات المساعدة فعل ذلك بجعل المورثات على الدنا أكثر عرضة للـSTATs وبتوظيف بروتينات ضرورية لانتساخ المورثات. يظهر التقاطع بين STATs والمنشطات المساعدة عبر سلاسل التقاطع (TADs) على STATs. يمكن لهذه الأخيرة أيضاً التقاطع مع أنزيمات histone ؛ [14] acetyltransferase(HATs) ، تضيف HATs مجموعات أستيل إلى نهايات الليزين على بروتينات مرتبطة بالدنا تُدعى الهيستونات. تزيل إضافة مجموعات أستيل الشحنة الإيجابية على نهايات الليزين وينتج عن ذلك روابط أضعف بين الهيستونات والدنا ما يجعل الدنا أكثر عرضة للـ STATs وما يزيد من انتساخ المورثات الهدف.
التكامل مع سبل ترميز أخرى
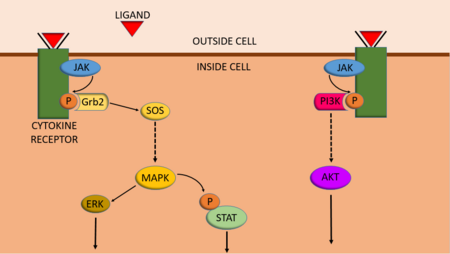
إيستطيع ترميز JAK-Stat التقاطع مع سبل ترميز خلوية أخرى مثل سبيل PI3K/AKT/mTOR. [15] عندما تتفعل JAKs وتفسفر نهايات التيروزين على المستقبلات، تستطيع البروتينات ذات سلاسل SH2 (مثلSTATs) ربط التيروزينات المفسفرة وتستطيع البروتينات أداء وظيفتها. يمتلك بروتين PI3K سلسلة SH2 مثل STATs ولذلك فهو قادر على الارتباط بهذه المستقبلات المفسفرة. بالنتيجة يمكن أن يفعل سبيل JAK-STAT سبيل ترميز PI3K/AKT/mTOR أيضاً.
يمكن أن يتكامل سبيل JAK-STAT أيضاً مع سبيل MAPK/ERK. أولاً، يمتلك بروتين مهم لترميز MAPK/ERK-يدعى Grb2- سلسلة SH2 وبذلك يمكنه الارتباط بالمستقبلات المفسفرة من قبل JAKs (بنفس طريقة PI3K). [15] يعمل Grb2 بعد ذلك ليسمح لسبيل MAPK/ERK بالتقدم. ثانياً، يستطيع بروتين مُفعل بسبيل MAPK/ERK ويُدعى MAPK(mitogen-activated protein kinase) فسفرة STATs، ما يمكن أن يزيد انتساخ المورثات بالـ STATs. على أية حال ورغم قدرة MAPK على زيادة الانتساخ المتواسط بـ STATs، تظهر دراسة أن فسفرة STAT3 بـ MAPK يمكن أن تقلل نشاط STAT3. [16]
أحد أمثلة تكامل ترميز JAK-STAT مع سبل أخرى هو ترميز مستقبل إنترلوكين 2 IL-2 في الخلايا التائية. تملك مستقبلات IL-2 سلاسل(غاما) المرتبطة بـ JAK3 التي تفسفر تيروزينات رئيسة على ذيل المستقبل. [17] توظف الفسفرة لاحقاً بروتين تكيف يُدعى Shc يُفعل سبيل MAPK/ERK وهذا يسهل تنظيم المورثا من قبل STAT5.
سبل الترميز البديلة
اُقترحت آلية بديلة لترميز JAK-STAT. في هذا النموذج، يمكن للكيناز الحاوي على سلسلة SH2 الارتباط بالتيروزينات المفسفرة على المستقبلات وفسفرة STATs مباشرة ما ينتج عنه تثاني STAT. [5] بعد ذلك وبخلاف الآلية التقليدية يمكن لـ STAT أن تُفسفر بالJAKs وبكينازات أخرى مرتبطة بالمستقبل. لذا إذا لم يعمل أحد الكينازات (JAKأو كيناز بديل حاوي على SH2)، فيمكن أن يظهر الترميز رغم ذلك عبر نشاط الكينازات الأخرى. وقد تم ذلك تجريبيا. [18]
دور في ترميز مستقبلات السيتوكين
نظراً لارتباط كثير من JAKs بمستقبلات السيتوكين، يؤدي سبيل ترميز JAK-STAT دوراُ رئيساً في ترميز مستقبل السيتوكين. بما أن السيتوكينات مواد تنتجها الخلايا المناعية ويمكنها التأثير على فعالية الخلايا المجاورة، فإن آثار JAK-STAT غالباً ما تُرى بكثافة في خلايا الجهاز المناعي. على سبيل المثال، تفعيل JAK3 استجابة لـIL-2 ضروري لتطوير الخلايا اللمفاوية ووظيفتها. [19] تظهر دراسة أن JAK1 ضروري لترميز مستقبلات السيتوكينات IFNγ وIL-2 وIL-4 وIL-10. [20]
يمكن لسبيل JAK-STAT في ترميز مستقبل السيتوكين أن يفعل STATs التي يمكن أن ترتبط بالدنا وتسمح بانتساخ الجينات المرتبطة بانقسام الخلايا المناعية وحياتها وتنشيطها وتوظيفها. مثلاً، قد يمكّن STAT1 انتساخ جينات تثبط انقسام الخلية وتفعل الالتهاب. [2] كما يمكن لـ STAT4 أن يفعل الخلايا القاتلة الطبيعية NK cells ويمكن لـ STAT5 أن يحرض تشكيل خلايا الدم البيضاء. [21] استجابة للسيتوكينات يمكن لترميز JAK-STAT أن يحفز STAT6 القادر على تحريض تكاثر الخلايا البائية، ونجاة الخلايا المناعية وإنتاج الجسم الضدي المدعى IgE.
الدور في التطوير
يؤدي ترميز JAK-STAT دوراً مهماً في تطور الحيوانات. يستطيع السبيل تحريض انقسام خلايا الدم وتمايزها(عملية تخصص الخلية). [22] في بعض الذباب ذي مورثات JAK الشاذة، قد يظهر انقسام كبير في الخلايا الدموية، ما قد ينجم عنه سرطان دموي. [23] ارتبط ترميز JAK-STAT أيضًا بالانقسام المفرط لخلايا الدم البيضاء في البشر والفئران.
سبيل الترميز مهم أيضاً في تطور عين ذبابة الفواكه (Drosophila melanogaster). عند ظهور في المورثات المرمزة لـ JAKs قد تعجز بعض خلايا العين عن الانقسام وتعجز خلايا أخرى مثل الخلايا المستقبلة للضوء عن التطور بشكل صحيح. [22]
تسبب الإزالة الكاملة لـ JAK وSTAT الموت في أجنة Drosophila في حين قد تسبب الطفرات في مورثات JAKs وSTATs تشوهات في أجساد الذباب وخاصة عيوباً في تشكيل القطع الجسدية. [22] تقول إحدى النظريات في سبب ظهور هذه التشوهات عند التعديل على JAK-STAT بأن STATs قد ترتبط مباشرة بالدنا وتحرض انتساخ المورثات المتدخلة في تشكيل القطع الجسدية ولذلك وبظهور JAKs أو STATs تعاني الذبابات من تشوهات في التقطع. [24] حُددت مواقع رابطة لـ STATs على أحد هذه المورثات، تُدعى even-skipped(eve) ما يدعم هذه النظرية. [25] من بين كل شرائط القطع المتأثرة بطفرات JAK أو STAT، الشريط الخامس هو الأكثر تأثراً، ما زالت الأسباب الجزيئية الدقيقة خلف هذا الأمر مجهولة.
التنظيم
نظراً لأهمية سبيل ترميز JAK-STAT وخاصة في ترميز السيتوكين، هناك عدد من الآليات التي تمتلكها الخلايا لتنظيم حجم الترميز الحادث. ثلاث مجموعات رئيسة من البروتينات التي تستخدمها الخلايا لتنظيم سبيل الترميز هذا هي مثبطات البروتين من STAT مفعلة، [26] فوسفاتاز التيروزين البروتيني (PTPs) [27] ومثبطات ترميز السيتوكين (SOCS). [28]
مثبطات البروتين من الSTATs المفعلة (PIAS)

PIAS هي عائلة بروتينية مكونة من أربعة بروتينات: PIAS1 و PIAS3 و PIASx و PIASγ . [29] تضيف البروتينات علامة، تسمى SUMO (معدل صغير يشبه ubiquitin) ، على بروتينات أخرى - مثل JAKs و STATs ، وتعديل وظيفتها. إضافة مجموعة SUMO على STAT1من قبل PIAS1 أظهرت منع تفعيل الجينات بـ STAT1. [30] أوضحت دراسات أخرى أن إضافة مجموعة SUMO إلى STATs قد تمنع فسفرة التيروزينات على STATs ما يمنع تثانيها ويثبط سبيل JAK-STAT. [31] وقد ثبت أن PIASγ يمنع STAT1 من العمل. [32] قد تعمل بروتينات PIAS أيضاً عبر منع STATs من الارتباط بالدنا (وبالتالي منع تفعيل الدنا) وبتوظيف بروتينات تُدعى histone deacetylase أو نازعة أستيل الهيستون (HDACs) ما يخفض درجة التعبير المورثي.
أنزيمات Protein tyrosine phosphatases(PTPs)
بما أن إضافة مجموعات فوسفات على التيروزينات جزء مهم من آلية عمل سبيل ترميز JAK-STAT فإن نزع مجموعات الفوسفات هذه قد يثبط الترميز. PTPs هي تيروزين ديفوسفتاز وهي قادرة على نزع هذه الفوسفات ومنع الترميز. ثلاثة PTPs رئيسة هي SHP-1 وSHP-2 و CD45. [33]
- SHP-1 . يتم التعبير عن SHP-1 بشكل أساسي في خلايا الدم . [34] تحتوي على سلسلتي SH2 وسلسلة محفزة (منطقة من البروتين تؤدي وظيفته الرئيسة)- تحتوي السلسلة المحفزة على سلسلة الحمض الأميني VHCSAGIGRTG (سلسلة نموذجية PTPs). [35] ككل PTPs، بعض بنى الحموض الأمينية أساسية لوظيفتها:حموض السيستين والأرجنين والغلوتامين الأمينية المصونة، وحلقة مكونة من حموض التريبتوفان والبرولين والأسبارتات (WAD). عندما تكون SHP-1 غير فعالة، تندمج سلاسل SH2 مع السلسلة المحفزة وبذلك يكون الفوسفاتاز عاجزاً عن العمل. عند تفعيل SHP-1 تبتعد سلاسل SH2 عند السلسلة المحفزة كاشفة الموقع المحفز ما يسمح بنشاط الفوسفاتاز. يستطيع SHP-1 بعد ذلك الارتباط وإزالة مجموعة الفوسفات من JAKs المرتبطة بالمستقبلات، ما يمنع الفسفرة الضرورية لاستمرار سبيل الترميز.
أحد أمثلة ذلك يُرى في سبيل ترميز JAK-STAT المتواسط بمستقبل الإيرثروبويتين(EpoR). يرتبط هنا SHP-1 مباشرة بنهاية تيروزين (في الموقع 429) على EpoR وتزيل مجموعات الفوسفات من المستقبل المرتبط بـ JAK2. [36] قدرة SHP-1 على التنظيم السلبي لسبيل JAK-STAT لوحظت أيضاً في تجارب على فئران تفتقد SHP-1. [37] تعاني هذه الفئران من مميزات الأمراض المناعية الذاتية وتظهر قدراً عالياً من تكاثر الخلايا، وهي مميزات نموذجية لدرجة عالية شاذة من ترميز JAK-STAT. بالإضافة لذلك، رُبطت إضافة مجموعة الميثيل إلى مورثة SHP-1 (ما يقلل من كمية SHP-1 المُنتج) باللمفوما(نوع من سرطان الدم). [38]
لكن قد يحرّض SHP-1 ترميز JAK-STAT. وجدت دراسة أُجريت عام 1997 أن SHP-1 يُحتمل أن يسمح بقدر كبير من تفعيل STAT على عكس نشاط STAT. [39] ما زال الفهم الجزيئي الدقيق لكيفية تفعيل SHP-1 وتثبيطه لسبيل الترميز مجهولاً. [33]
- يمتلك SHP-2 بنية مشابهة كثيراً لـ SPH-1 لكن وبخلاف SHP-1، يُنتج SHP-2 في كثير من أنماط الخلايا وليس في خلايا الدم فقط. [40] يمتلك البشر بروتيني SHP-2 كل منهما مكون من 593 و597 حمض أميني. [35] يبدو أن سلاسل SH2 في SHP-2 تؤدي دوراً مهماً في التحكم بنشاط SHP-2. ترتبط إحدى سلاسل SH2 بالسلسلة المحفزة في SHP-2 لتمنع عمل SHP-2. [33] ثم وعندما يرتبط بروتين مفسفر التيروزين تتغير عمليات سلسلة SH2 وتتفعل SHP-2. يستطيع SHP-2 بعدها إزالة مجموعات الفوسفات من JAKs وSTATs والمستقبلات نفسها وبذلك ومثل SPH-1 يستطيع منع الفسفرة اللازمة لاستمرار السبيل وبالتالي يثبط سبيل JAK-STAT. SHP-2 قادر مثل SHP-1 على إزالة مجموعات الفوسفات هذه عبر عمل السيستين والأرجنين والغلوتامين المحفوظة وحلقة WPD.
لُوحظ التنظيم السلبي لـSHP-2 في عدد من التجارب، أحد الأمثلة كان عند استكشاف ترميز JAK1/STAT1 حيث يستطيع SHP-2 إزالة مجموعات فوسفات من البروتينات في السبيل مثل STAT1. [41] بطريقة مماثلة، أظهر SHP-2 قدرة على تقليل الترميز الحاوي على بروتينات STAT3 وSTAT5 بنزع مجموعات الفوسفات. [42] [43]
يُعتقد أن SHP-2 كحال SHP-1 يحفز ترميز JAK-STAT في بعض الحالات ويثبط الترميز أيضاً. تظهر دراسة مثلاً أن SHP-2 قد يحفز نشاط STAT5 بدلاً من تقليله. [44] تقترح دراسات أخرى أيضاً أن SHP-2 قد يزيد نشاط JAK2 ويحرض ترميز JAK2/STAT. [45] لا تزال كيفية قدرة SHP2 على تثبيط وتحفي ترميز JAK-STAT مجهولة في سبيل JAK2/STAT5: تقترح إحدى النظريات أن SHP-2 قد يحرض تفعيل JAK2 لكنه يثبط STAT5 بإزالة مجموعات الفوسفات منه. [33]
- CD45 . يتم إنتاج CD45 بشكل رئيسي في خلايا الدم. [4] في البشر ثبت أنه قادر على العمل على JAK1 و JAK3 ، [46] بينما في الفئران، CD45 قادر على العمل على جميع JAKs. [47] تشير إحدى الدراسات إلى أن CD45 يمكن أن يقلل من مقدار الوقت الذي تنشط فيه إشارات JAK-STAT. لا تزال التفاصيل الدقيقة لكيفية عمل وظائف CD45 غير معروفة. [33]
كابحات ترميز السيتوكين (SOCS)
هناك ثمانية بروتينات من عائلة SOCS: بروتين مفعل السيتوكين الحاوي على سلسلة SH2 (CISH) وSOCS1 وSOCS2 وSOCS3 وSOCS4 وSOCS5 وSOCS6 وSOCS7، يمتلك كل بروتين سلسلة SH2 ومنطقة من أربعين حمض أميني تُدعى صندوق. [48] قد يندمج صندوق SOCS مع عدد من البروتينات ليشكل معقداً بروتينياً ويستطيع هذا المعقد أن يتسبب بتفكك JAKs والمستقبلات ذاتها ويثبط بالتالي ترميز JAK-STAT. [4] يؤدي معقد البروتين ذلك عبر السماح لواسم يُدعى يوبيكويتين بأن يُضاف للبروتينات في عملية تُدعى ubiquitination والتي تشير للبروتين بالتحطم. [49] تنتقل البروتينات مثل JAKs والمستقبلات بعدها إلى حجرة في الخلية تُدعى بروتيازوم تنفذ تحطيم البروتينات.
تعمل SOCS أيضاً بالارتباط ببروتينات عاملة في ترميز JAK-STAT ومنع نشاطها. مثلاً، ترتبط سلسلة SH2 من SOCS1 بتيروزين في حلقة التفعيل من JAKs ما يمنع JAKs من فسفرة بعضها البعض. [4] ترتبط سلاسل SH2 من SOCS2 وSOCS3 وCIS مباشرة بالمستقبلات ذاتها. [49] يستطيع SOCS1 وSOCS3 أيضاً منع ترميز JAK-STAT بالارتباط بـ JAKs باستخدام قطع تُدعى kinase inhibitory regions(KIRs) مناطق تثبيط الكيناز وإيقاف ارتباط JAKs ببروتينات أخرى. [50] طريقة عمل SOCS الأخرى الدقيقة أكثر غموضاً.
الأهمية السريرية
بما أن سبيل JAK-STAT يؤدي دوراً رئيساً في كثير من العمليات الأساسية مثل الموت الخلوي المبرمج والالتهاب، فقد تؤدي البروتينات الشاذة إلى عدد من الأمراض. قد تسبب الشذوذات في ترميز JAK-STAT سرطانات وأمراض تؤثر على الجهاز المناعي مثل عوز المناعة المشترك الشديد. [51]
الأمراض المتعلقة بالجهاز المناعي
قد يُستخدم JAK3 لترميز IL-2 وIL-4 وIL-15 وIL-21 (وغيرها من السيتوكينات) ولهذا قد يعاني المرضى ذوو الطفرات في مورثة JAK3 غالباً من اضطرابات تخص كثيراً من جوانب الجهاز المناعي. [52] [53] مثلاً، يسبب JAK3 الشاذ عوز المناعة المشترك الشديد ما يسبب عدم امتلاك المرضى للخلايا القاتلة الطبيعية والخلايا البائية والخلايا التائية وهذا ما يجعل المعانين منه معرضين للإنتان. ظهرت تسبب طفرات البروتين STAT5 والذي يمكن أن يرمز JAK3 باضطرابات مناعية ذاتية. [54]
اُقترح أن المرضى ذوي طفرات في STAT1 وSTAT2 أكثر عرضة للإصابة بإنتانات جرثومية وفيروسية. [55] أكما ارتبطت طفرات STAT4 بالداء الرثياني وطفرات STAT6 بالربو. [56] [57]
قد يعاني المرضى ذوو سبيل ترميز Jak-STAT شاذ أيضاً من أمراض جلدية. ارتبط على سبيل المثال وجود مستقبلات شاذة للسيتوكين وزيادة تعبير STAT3 بالصداف (مرض مناعي ذاتي يتميز بجلد أحمر رقيق متقشر). [53] يؤدي STAT3 دوراً مهماً في الصدفية إذ يمكن أن يتحكم STAT3 بإنتاج مستقبلات IL-23 ويمكن لهذا الأخير أن يساعد على تطوير خلايا Th17، ويمكن أن تثير الخلايا Th17 حدوث الصدفية. [58] بما أن كثيراً من السيتوكينات تعمل عبر عامل انتساخ STAT3 ، يؤدي هذا الأخير دوراً هاماً في صيانة مناعة الجلد. علاوة على ذلك، بما أن المرضى ذوي الطفرات في مورثة JAK3 لا يملكون خلايا تائية وبائية وقاتلة طبيعية سليمة فإنهم أكثر عرضة للإصابة بالإنتانات الجلدية.
السرطان
يشمل السرطان نمواً خلوياً شاذا وخارجاً عن السيطرة في جزء من الجسد. لذا وبما أن ترميز JAK-STAT يمكن أن يسمح بانتساخ مورثات ذات دور في انقسام الخلية، فإن أثراً محتملاً لترميز JAK-STAT الزائد هو تشكل السرطان. رُبطت الدرجات المرتفعة من تفعيل STAT بالسرطان، الدرجات المرتفعة من تفعيل STAT3 وSTAT5 بالتحديد هي الأكثر ارتباطاً بالأورام الأخطر. [59] رُبطت مثلاً زيادة نشاط STAT3 بزيادة احتمالية عودة الميلانوما(سرطان جلدي) بعد علاجه ورُبطت الدرجات العالية من نشاط STAT5 بزيادة احتمال موت مرضى سرطان البروستات. [60] قد يرتبط ترميز JAK-STAT بتطور سرطان الثدي. قد يحرض ترميزJAK-STAT في الغدد الثديية( الموجودي في الثدي) الانقسام الخلوي ويقلل الموت الخلوي المبرمج خلال الحمل والبلوغ وبالتالي فإنه قد يسبب تشكل السرطان عند زيادة تفعيله. [61] يؤدي نشاط STAT3 دوراً رئيساً في هذه العملية بما أنه قد يسمح بانتساخ مورثات مثل BCL2 وc-Myc المرتبطة بالانقسام الخلوي.
يمكن أن تؤدي الطفرات في JAK2 إلى سرطان الدم والأورام اللمفاوية . [5] على وجه التحديد، يُقترح أن تكون الطفرات في exons 12 و 13 و 14 و 15 من جين JAK2 عامل خطر في الإصابة بسرطان الغدد الليمفاوية أو سرطان الدم. علاوة على ذلك قد تسبب طفرات في STAT3 وSTAT5 زيادة ترميز JAK-STAT في الخلايا القاتلة الطبيعية والتائية ما قد يحرض تكاثراً كبيراً في هذه الخلايا وزيادة في احتتمال تطور اللوكيميا. [61] قد يشذ أيضاً سبيل ترميز JAK-STAT المتواسط بالإرثروبوتين، الذي يسمح عادة بتطور خلايا الدم الحمراء، في مرضى اللوكيميا. [62]
العلاجات
بما أن الزيادة في ترميز JAK-STAT مسؤولة عن بعض السرطانات والأمراض المناعية فإن مثبطات JAK اُقترحت كعقارات علاجية. مثلاً، لعلاج بعض أشكال اللوكيميا فإن استهداف وتثبيط JAKs قد يزيل آثار ترميز الإرثروبوتين وربما يمنع تطور اللوكيميا. [62] أحد مثبطات JAK هو Ruxolitinib المستخدم كمثبط JAK2. [59] تُطور مثبطات STAT وتستهدف الكثير من المثبطات STAT3. [61] سُجل أن العلاجات التي تستهدف STAT3 قد تحسن فرص نجاة مرضى السرطان. يُستخدم عقار آخر يدعى Toacitinib لعلاج الصداف والداء الرثياني. [51]
المراجع
- "A road map for those who don't know JAK-STAT". Science. 296 (5573): 1653–5. 2002. doi:10.1126/science.1071545. PMID 12040185.
- Schindler, Christian; Levy, David E.; Decker, Thomas (2007). "JAK-STAT Signaling: From Interferons to Cytokines". Journal of Biological Chemistry. 282 (28): 20059–20063. doi:10.1074/jbc.R700016200.
- Kaneko, Tomonori; Joshi, Rakesh; Feller, Stephan M; Li, Shawn SC (2012). "Phosphotyrosine recognition domains: the typical, the atypical and the versatile". Cell Communication and Signaling. 10 (1): 32. doi:10.1186/1478-811X-10-32. PMID 23134684.
- Kiu, Hiu; Nicholson, Sandra E. (2012). "Biology and significance of the JAK/STAT signalling pathways". Growth Factors. 30 (2): 88–106. doi:10.3109/08977194.2012.660936. PMID 22339650.
- Jatiani, S. S.; Baker, S. J.; Silverman, L. R.; Reddy, E. P. (2011). "JAK/STAT Pathways in Cytokine Signaling and Myeloproliferative Disorders: Approaches for Targeted Therapies". Genes & Cancer. 1 (10): 979–993. doi:10.1177/1947601910397187. PMID 21442038.
- Reich, Nancy C; Rout, M. P. (2014). "STATs get their move on". JAK-STAT. 2 (4): 27080. doi:10.4161/jkst.27080. PMID 24470978.
- Liu, L.; McBride, K. M.; Reich, N. C. (2005). "STAT3 nuclear import is independent of tyrosine phosphorylation and mediated by importin- 3". Proceedings of the National Academy of Sciences. 102 (23): 8150–8155. doi:10.1073/pnas.0501643102. PMID 15919823.
- Yang, J.; Huang, J.; Dasgupta, M.; Sears, N.; Miyagi, M.; Wang, B.; Chance, M. R.; Chen, X.; Du, Y. (2010). "Reversible methylation of promoter-bound STAT3 by histone-modifying enzymes". Proceedings of the National Academy of Sciences. 107 (50): 21499–21504. doi:10.1073/pnas.1016147107. PMID 21098664.
- Stark, George R.; Darnell, James E. (2012). "The JAK-STAT Pathway at Twenty". Immunity. 36 (4): 503–514. doi:10.1016/j.immuni.2012.03.013. PMID 22520844.
- Zhuang, Shougang (2013). "Regulation of STAT signaling by acetylation". Cellular Signalling. 25 (9): 1924–1931. doi:10.1016/j.cellsig.2013.05.007. PMID 23707527.
- Ma, L.; Gao, J.-s.; Guan, Y.; Shi, X.; Zhang, H.; Ayrapetov, M. K.; Zhang, Z.; Xu, L.; Hyun, Y.-M. (2010). "Acetylation modulates prolactin receptor dimerization". Proceedings of the National Academy of Sciences. 107 (45): 19314–19319. doi:10.1073/pnas.1010253107. PMID 20962278.
- Shen, Y.; Schlessinger, K.; Zhu, X.; Meffre, E.; Quimby, F.; Levy, D. E.; Darnell, J. E. (2003). "Essential Role of STAT3 in Postnatal Survival and Growth Revealed by Mice Lacking STAT3 Serine 727 Phosphorylation". Molecular and Cellular Biology. 24 (1): 407–419. doi:10.1128/MCB.24.1.407-419.2004. PMID 14673173.
- Decker, Thomas; Kovarik, Pavel (2000). "Serine phosphorylation of STATs". Oncogene. 19 (21): 2628–2637. doi:10.1038/sj.onc.1203481.
- Paulson, Matthew; Press, Carolyn; Smith, Eric; Tanese, Naoko; Levy, David E. (2002). "IFN-Stimulated transcription through a TBP-free acetyltransferase complex escapes viral shutoff". Nature Cell Biology. 4 (2): 140–147. doi:10.1038/ncb747.
- Rawlings, Jason S.; Rosler, Kristin M.; Harrison, Douglas A. (2004). "The JAK/STAT signaling pathway". Journal of Cell Science. 117 (8): 1281–1283. doi:10.1242/jcs.00963. PMID 15020666.
- Jain, Neeraj; Zhang, Tong; Fong, Siok Lyn; Lim, Cheh Peng; Cao, Xinmin (1998). "Repression of Stat3 activity by activation of mitogen-activated protein kinase (MAPK)". Oncogene. 17 (24): 3157–3167. doi:10.1038/sj.onc.1202238. PMID 9872331.
- Malek, Thomas R.; Castro, Iris (2010). "Interleukin-2 Receptor Signaling: At the Interface between Tolerance and Immunity". Immunity. 33 (2): 153–165. doi:10.1016/j.immuni.2010.08.004. PMID 20732639.
- Sen, B.; Saigal, B.; Parikh, N.; Gallick, G.; Johnson, F. M. (2009). "Sustained Src Inhibition Results in Signal Transducer and Activator of Transcription 3 (STAT3) Activation and Cancer Cell Survival via Altered Janus-Activated Kinase-STAT3 Binding". Cancer Research. 69 (5): 1958–1965. doi:10.1158/0008-5472.CAN-08-2944.
- Smith, Geoffrey A; Uchida, Kenji; Weiss, Arthur; Taunton, Jack (2016). "Essential biphasic role for JAK3 catalytic activity in IL-2 receptor signaling". Nature Chemical Biology. 12 (5): 373–379. doi:10.1038/nchembio.2056.
- Rodig, Scott J; Meraz, Marco A; White, J.Michael; Lampe, Pat A; Riley, Joan K; Arthur, Cora D; King, Kathleen L; Sheehan, Kathleen C.F; Yin, Li (1998). "Disruption of the Jak1 Gene Demonstrates Obligatory and Nonredundant Roles of the Jaks in Cytokine-Induced Biologic Responses". Cell. 93 (3): 373–383. doi:10.1016/S0092-8674(00)81166-6. PMID 9590172.
- Grebien, F.; Kerenyi, M. A.; Kovacic, B.; Kolbe, T.; Becker, V.; Dolznig, H.; Pfeffer, K.; Klingmuller, U.; Muller, M. (2008). "Stat5 activation enables erythropoiesis in the absence of EpoR and Jak2". Blood. 111 (9): 4511–4522. doi:10.1182/blood-2007-07-102848.
- Luo, Hong; Dearolf, Charles R. (2001). "The JAK/STAT pathway andDrosophila development". BioEssays. 23 (12): 1138–1147. doi:10.1002/bies.10016.
- Luo, H; Rose, P; Barber, D; Hanratty, W P; Lee, S; Roberts, T M; D'Andrea, A D; Dearolf, C R (1997). "Mutation in the Jak kinase JH2 domain hyperactivates Drosophila and mammalian Jak-Stat pathways". Molecular and Cellular Biology. 17 (3): 1562–1571. doi:10.1128/MCB.17.3.1562.
- Binari, R; Perrimon, N (1994). "Stripe-specific regulation of pair-rule genes by hopscotch, a putative Jak family tyrosine kinase in Drosophila". Genes & Development. 8 (3): 300–312. doi:10.1101/gad.8.3.300.
- Yan, Riqiang; Small, Stephen; Desplan, Claude; Dearolf, Charles R; Darnell, James E; Roberts, T M; D'Andrea, A D; Dearolf, C R (1996). "Identification of a Stat Gene That Functions in Drosophila Development". Cell. 84 (3): 421–430. doi:10.1016/S0092-8674(00)81287-8.
- "Regulation of cytokine signaling pathways by PIAS proteins". Cell Research. 16 (2): 196–202. 2006. doi:10.1038/sj.cr.7310027. PMID 16474434. 16474434.
- Henenstreit, D.; Horeks-Hoeck, J.; Duschl, A. (2005). "JAK/STAT-dependent gene regulation by cytokines". Drug News & Perspectives. 18 (4): 243. doi:10.1358/dnp.2005.18.4.908658. PMID 16034480.
- "SOCS proteins: negative regulators of cytokine signaling". Stem Cells. 19 (5): 378–87. 2001. doi:10.1634/stemcells.19-5-378. PMID 11553846.
- Shuai, Ke; Liu, Bin; Zhang, Di; Cui, Yan; Zhou, Jinlian; Cui, Sheng (2005). "Regulation of gene-activation pathways by PIAS proteins in the immune system". Nature Reviews Immunology. 5 (8): 593–605. doi:10.1038/nri1667. PMID 16056253.
- Ungureanu, D.; Vanhatupa, S.; Grönholm, J.; Palvimo, J.; Silvennoinen, O. (2005). "SUMO-1 conjugation selectively modulates STAT1-mediated gene responses". Blood. 106 (1): 224–226. doi:10.1182/blood-2004-11-4514.
- Droescher, Mathias; Begitt, Andreas; Marg, Andreas; Zacharias, Martin; Vinkemeier, Uwe (2011). "Cytokine-induced Paracrystals Prolong the Activity of Signal Transducers and Activators of Transcription (STAT) and Provide a Model for the Regulation of Protein Solubility by Small Ubiquitin-like Modifier (SUMO)". Journal of Biological Chemistry. 286 (21): 18731–18746. doi:10.1074/jbc.M111.235978.
- Liu, B.; Gross, M.; ten Hoeve, J.; Shuai, K. (2001). "A transcriptional corepressor of Stat1 with an essential LXXLL signature motif". Proceedings of the National Academy of Sciences. 98 (6): 3203–3207. doi:10.1073/pnas.051489598. PMID 11248056.
- Xu, Dan; Qu, Cheng-Kui (2008). "Protein tyrosine phosphatases in the JAK/STAT pathway". Frontiers in Bioscience. 13 (1): 4925–4932. doi:10.2741/3051.
- Yi, T L; Cleveland, J L; Ihle, J N (1992). "Protein tyrosine phosphatase containing SH2 domains: characterization, preferential expression in hematopoietic cells, and localization to human chromosome 12p12-p13". Molecular and Cellular Biology. 12 (2): 836–846. doi:10.1128/MCB.12.2.836.
- M. Scott, Latanya; R. Lawrence, Harshani; M. Sebti, Said; J. Lawrence, Nicholas; Wu, Jie (2010). "Targeting Protein Tyrosine Phosphatases for Anticancer Drug Discovery". Current Pharmaceutical Design. 16 (16): 1843–1862. doi:10.2174/138161210791209027.
- Bone, Heather; Dechert, Ute; Jirik, Frank; Schrader, John W.; Welham, Melanie J. (1997). "SHP1 and SHP2 Protein-tyrosine Phosphatases Associate with βc after Interleukin-3-induced Receptor Tyrosine Phosphorylation". Journal of Biological Chemistry. 272 (22): 14470–14476. doi:10.1074/jbc.272.22.14470.
- Lyons, Bonnie L; Lynes, Michael A; Burzenski, Lisa; Joliat, Melissa J; Hadjout, Nacima; Shultz, Leonard D (2003). "Mechanisms of anemia in SHP-1 protein tyrosine phosphatase-deficient "viable motheaten" mice". Experimental Hematology. 31 (3): 234–243. doi:10.1016/S0301-472X(02)01031-7.
- Johan, M. F.; Bowen, D. T.; Frew, M. E.; Goodeve, A. C.; Reilly, J. T. (2005). "Aberrant methylation of the negative regulators RASSFIA, SHP-1 and SOCS-1 in myelodysplastic syndromes and acute myeloid leukaemia". British Journal of Haematology. 129 (1): 60–65. doi:10.1111/j.1365-2141.2005.05412.x.
- You, Min; Zhao, Zhizhuang (1997). "Positive Effects of SH2 Domain-containing Tyrosine Phosphatase SHP-1 on Epidermal Growth Factor- and Interferon-γ-stimulated Activation of STAT Transcription Factors in HeLa Cells". Journal of Biological Chemistry. 272 (37): 23376–23381. doi:10.1074/jbc.272.37.23376.
- Neel, Benjamin G.; Gu, Haihua; Pao, Lily (2003). "The 'Shp'ing news: SH2 domain-containing tyrosine phosphatases in cell signaling". Trends in Biochemical Sciences. 28 (6): 284–293. doi:10.1016/S0968-0004(03)00091-4. PMID 12826400.
- Wu, Tong R.; Hong, Y. Kate; Wang, Xu-Dong; Ling, Mike Y.; Dragoi, Ana M.; Chung, Alicia S.; Campbell, Andrew G.; Han, Zhi-Yong; Feng, Gen-Sheng (2002). "SHP-2 Is a Dual-specificity Phosphatase Involved in Stat1 Dephosphorylation at Both Tyrosine and Serine Residues in Nuclei". Journal of Biological Chemistry. 277 (49): 47572–47580. doi:10.1074/jbc.M207536200.
- Chen, Yuhong; Wen, Renren; Yang, Shoua; Schuman, James; Zhang, Eric E.; Yi, Taolin; Feng, Gen-Sheng; Wang, Demin (2003). "Identification of Shp-2 as a Stat5A Phosphatase". Journal of Biological Chemistry. 278 (19): 16520–16527. doi:10.1074/jbc.M210572200.
- Zhang, E. E.; Chapeau, E.; Hagihara, K.; Feng, G.-S. (2004). "Neuronal Shp2 tyrosine phosphatase controls energy balance and metabolism". Proceedings of the National Academy of Sciences. 101 (45): 16064–16069. doi:10.1073/pnas.0405041101.
- Ke, Yuehai; Lesperance, Jacqueline; Zhang, Eric E.; Bard-Chapeau, Emilie A.; Oshima, Robert G.; Muller, William J.; Feng, Gen-Sheng (2006). "Conditional Deletion of Shp2 in the Mammary Gland Leads to Impaired Lobulo-alveolar Outgrowth and Attenuated Stat5 Activation". Journal of Biological Chemistry. 281 (45): 34374–34380. doi:10.1074/jbc.M607325200.
- Yu, Wen-Mei; Hawley, Teresa S; Hawley, Robert G; Qu, Cheng-Kui (2003). "Catalytic-dependent and -independent roles of SHP-2 tyrosine phosphatase in interleukin-3 signaling". Oncogene. 22 (38): 5995–6004. doi:10.1038/sj.onc.1206846.
- Yamada, Takechiyo; Zhu, Daocheng; Saxon, Andrew; Zhang, Ke (2002). "CD45 Controls Interleukin-4-mediated IgE Class Switch Recombination in Human B Cells through Its Function as a Janus Kinase Phosphatase". Journal of Biological Chemistry. 277 (32): 28830–28835. doi:10.1074/jbc.M201781200.
- Irie-Sasaki, Junko; Sasaki, Takehiko; Matsumoto, Wataru; Opavsky, Anne; Cheng, Mary; Welstead, Grant; Griffiths, Emily; Krawczyk, Connie; Richardson, Christopher D. (2001). "CD45 is a JAK phosphatase and negatively regulates cytokine receptor signalling". Nature. 409 (6818): 349–354. doi:10.1038/35053086.
- Alexander, Warren S.; Hilton, Douglas J. (2004). "The role of suppressors of cytokine signaling (SOCS) proteins in regulation of the immune response". Annual Review of Immunology. 22 (1): 503–529. doi:10.1146/annurev.immunol.22.091003.090312.
- Tamiya, T.; Kashiwagi, I.; Takahashi, R.; Yasukawa, H.; Yoshimura, A. (2011). "Suppressors of Cytokine Signaling (SOCS) Proteins and JAK/STAT Pathways: Regulation of T-Cell Inflammation by SOCS1 and SOCS3". Arteriosclerosis, Thrombosis, and Vascular Biology. 31 (5): 980–985. doi:10.1161/ATVBAHA.110.207464.
- Kershaw, Nadia J.; Murphy, James M.; Lucet, Isabelle S.; Nicola, Nicos A.; Babon, Jeffrey J. (2013). "Regulation of Janus kinases by SOCS proteins". Biochemical Society Transactions. 41 (4): 1042–1047. doi:10.1042/BST20130077.
- Villarino, Alejandro V.; Kanno, Yuka; Ferdinand, John R.; O’Shea, John J. (2015). "Mechanisms of Jak/STAT Signaling in Immunity and Disease". The Journal of Immunology. 194 (1): 21–27. doi:10.4049/jimmunol.1401867.
- Pesu, Marko; Candotti, Fabio; Husa, Matthew; Hofmann, Sigrun R.; Notarangelo, Luigi D.; O'Shea, John J. (2005). "Jak3, severe combined immunodeficiency, and a new class of immunosuppressive drugs". Immunological Reviews. 203 (1): 127–142. doi:10.1111/j.0105-2896.2005.00220.x. PMID 15661026.
- Welsch, Katharina; Holstein, Julia; Laurence, Arian; Ghoreschi, Kamran (2017). "Targeting JAK/STAT signalling in inflammatory skin diseases with small molecule inhibitors". European Journal of Immunology. 47 (7): 1096–1107. doi:10.1002/eji.201646680.
- Casanova, Jean-Laurent; Holland, Steven M.; Notarangelo, Luigi D. (2012). "Inborn Errors of Human JAKs and STATs". Immunity. 36 (4): 515–528. doi:10.1016/j.immuni.2012.03.016.
- Au-Yeung, Nancy; Mandhana, Roli; Horvath, Curt M (2014). "Transcriptional regulation by STAT1 and STAT2 in the interferon JAK-STAT pathway". JAK-STAT. 2 (3): 23931. doi:10.4161/jkst.23931. PMID 24069549.
- Remmers, Elaine F.; Plenge, Robert M.; Lee, Annette T.; Graham, Robert R.; Hom, Geoffrey; Behrens, Timothy W.; de Bakker, Paul I.W.; Le, Julie M.; Lee, Hye-Soon (2007). "STAT4 and the Risk of Rheumatoid Arthritis and Systemic Lupus Erythematosus". New England Journal of Medicine. 357 (10): 977–986. doi:10.1056/NEJMoa073003. PMID 17804842.
- Vercelli, Donata (2008). "Discovering susceptibility genes for asthma and allergy". Nature Reviews Immunology. 8 (3): 169–182. doi:10.1038/nri2257.
- Ghoreschi, Kamran; Laurence, Arian; Yang, Xiang-Ping; Hirahara, Kiyoshi; O'Shea, John J. (2011). "T helper 17 cell heterogeneity and pathogenicity in autoimmune disease". Trends in Immunology. 32 (9): 395–401. doi:10.1016/j.it.2011.06.007. PMID 21782512.
- Thomas, S J; Snowden, J A; Zeidler, M P; Danson, S J (2015). "The role of JAK/STAT signalling in the pathogenesis, prognosis and treatment of solid tumours". British Journal of Cancer. 113 (3): 365–371. doi:10.1038/bjc.2015.233.
- Messina, Jane L.; Yu, Hua; Riker, Adam I.; Munster, Pamela N.; Jove, Richard L.; Daud, Adil I. (2008). "Activated Stat-3 in Melanoma". Cancer Control. 15 (3): 196–201. doi:10.1177/107327480801500302.
- Groner, Bernd; von Manstein, Viktoria (2017). "Jak Stat signaling and cancer: Opportunities, benefits and side effects of targeted inhibition". Molecular and Cellular Endocrinology. 451: 1–14. doi:10.1016/j.mce.2017.05.033.
- Kim, Jinkoo; Jung, Younghun; Sun, Hongli; Joseph, Jeena; Mishra, Anjali; Shiozawa, Yusuke; Wang, Jingcheng; Krebsbach, Paul H.; Taichman, Russell S. (2012). "Erythropoietin mediated bone formation is regulated by mTOR signaling". Journal of Cellular Biochemistry. 113 (1): 220–228. doi:10.1002/jcb.23347.
قراءة متعمقة
- Schroder K, Hertzog PJ, Ravasi T, Hume DA (February 2004). "Interferon-gamma: an overview of signals, mechanisms and functions". Journal of Leukocyte Biology. 75 (2): 163–89. doi:10.1189/jlb.0603252. PMID 14525967.
- O'Shea JJ, Gadina M, Schreiber RD (April 2002). "Cytokine signaling in 2002: new surprises in the Jak/Stat pathway". Cell. 109 Suppl (Suppl): S121-31. doi:10.1016/S0092-8674(02)00701-8. PMID 11983158.
