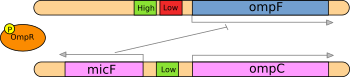
تنظيم ما بعد النسخ (Post-transcriptional regulation) هي مرحلة من تنظيم التعبير الجيني وتتكون من جميع الآليات التي يتم بواسطتها التأثير على التعبير الجيني على مستوى الرنا، أي بعد النسخ وقبل الترجمة.[2][3] يسمح تنظيم ما بعد النسخ للخلايا بالتأقلم مع التغيرات المناخية. لدى حقيقيات النوى جزيء الرنا الرسول الذي لا يخضع لتعديل ما بعد الترجمة يكون عمره أقصر نسبيا بسبب تواجد جزيئات الرناز وهي البروتينات التي تفكك الرنا.
التنظيم بواسطة الرنا الميكروي
تنظِّم جزيئات الرنا الميكروي التعبير عن ما يزيد عن 60% من الجينات المشفرة للبروتين في الجينوم البشري.[4] إذا تواجد الرنا الميكروي بكثرة فإن بإمكانه التصرف "كقاطعة" وتشغيل التعبير عن بعض الجينات أو إطفائه.[5] مع ذلك، التعبيرات الجينية المتحكم فيها بواسطة الرنا الميكروي لا تؤدي سوى إلى تغيرات متواضعة حوالي 1.5 إلى 4 مرات في مقدار التعبير عن الجينات المستهدفة.[5] عادة ما يثبط جزيء الرنا الميكروي المفرد عدة مئاتٍ من الجينات المستهدفة.[4][6] ويحدث التثبيط إما عبر إسكات ترجمة الرنا الرسول أو عبر تفكيكه بواسطة ترابط تكاملي يكون في الغالب مع تسلسلات في المنطقة 3' غير المترجمة من الرنا الرسول للجين المستهدف.[7] آلية إسكات أو تفكيك الرنا الرسول تتم بواسطة مركب ريسك (RISC ).
الأهمية
حصل هذا المجال من الدراسة على أهمية أكبر بسبب الدلائل المتزايدة على أن تنظيم ما بعد النسخ يلعب دورا أكبر مما اعتُقد سابقا. رغم أن البروتينات ذات النطاقات المرتبطة بالدنا أكثر وفرة من البروتينات ذات النطاقات المرتبطة بالرنا، أظهرت دراسة حديثة أنه أثناء تنشيط خلية تائية 55% من التغييرات المعتبرة في معدل حالة الاستقرار لم يكن لها تغيرات ذات صلة على معدل النسخ، ما يعني أنها كانت نتيجة لتنظيم الاستقرار وحده.[8]
فضلا عن ذلك، الرنا الموجود في النواة أكثر تعقيدا من نظيره الموجود في السيوتوبلازم: أزيد من 95% من الرنا (القواعد) المخلق بواسطة بوليميراز الرنا 2 لا يصل أبدا إلى السيتوبلازم. السبب الرئيسي لذلك هو إزالة الإنترونات والتي تشكل حوالي 80% من مجموع القواعد.[9] أظهرت بعض الدراسات أنه حتى بعد عملية المعالجة معدلات الرنا الرسول بين النواة والخلية مختلفة بشكل كبير جدا.[10]
علم الأحياء النمائي مصدر جيد لآليات التنظيم، لكن لصعوبات تقنية فإنه من السهل تحديد تسلسل عمليات عامل النسخ بدل التنظيم على مستوى الرنا. في الحقيقة، جينات مفتاحية مثل نانوس يُعرف بأنها ترتبط بالرنا، لكن في الغالب أهدافها غير معروفة.[11] رغم أن الرنا المرتبط بالبروتينات يمكن أن ينظم ما بعد النسخ كمية كبيرة من النسخوم، إلا أن استهداف جين واحد هو ما يهمُّ المجتمع العلمي لأسباب طبية. تدخل الرنا والرنا الميكروي مثالان على تنظيم ما بعد النسخ، حيث ينظمان تفكيك الرنا وتغيير بنية الكروماتين. تُستخدم العديد من التقنيات لدراسة تعديل ما بعد النسخ مثل رقاقة ريب ( الترسيب المناعي للرنا على رقاقة).[12]
دور الرنا الميكروي في السرطان
مراجع
- Mims, Cedric A.; Nash, Anthony; Stephen, John (2001). Mims' Pathogenesis of Infectious Disease (الطبعة 5th). Academic Press. . مؤرشف من الأصل في 15 ديسمبر 2019.
- Bruce Alberts; Alexander Johnson; Julian Lewis; Martin Raff; Keith Roberts; Peter Walter (2007). Molecular Biology of the Cell (الطبعة Fifth). Garland Science. صفحات 1268 pages. .
- Weaver, Robert J. (2007). "Part V: Post-transcriptional events". Molecular Biology. Boston: McGraw Hill Higher Education. .
- Friedman RC, Farh KK, Burge CB, Bartel DP (2009). "Most mammalian mRNAs are conserved targets of microRNAs". Genome Res. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC . PMID 18955434.
- Farazi TA, Spitzer JI, Morozov P, Tuschl T (2011). "miRNAs in human cancer". J. Pathol. 223 (2): 102–15. doi:10.1002/path.2806. PMC . PMID 21125669.
- Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM (2005). "Microarray analysis shows that some microRNAs downregulate large numbers of the target mRNAs". Nature. 433 (7027): 769–73. doi:10.1038/nature03315. PMID 15685193.
- Hu W, Coller J (2012). "What comes first: translational repression or mRNA degradation? The deepening mystery of microRNA function". Cell Res. 22 (9): 1322–4. doi:10.1038/cr.2012.80. PMC . PMID 22613951.
- Cheadle C, Fan J, Cho-Chung YS, Werner T, Ray J, Do L, Gorospe M, Becker KG (2005). "Control of gene expression during T cell activation: alternate regulation of mRNA transcription and mRNA stability". BMC Genomics. 6: 75. doi:10.1186/1471-2164-6-75. PMC . PMID 15907206.
- Jackson DA, Pombo A, Iborra F (2000). "The balance sheet for transcription: an analysis of nuclear RNA metabolism in mammalian cells". FASEB J. 14 (2): 242–54. doi:10.1096/fasebj.14.2.242. PMID 10657981. مؤرشف من الأصل في 17 يناير 2010.
- Schwanekamp JA, Sartor MA, Karyala S, Halbleib D, Medvedovic M, Tomlinson CR (2006). "Genome-wide analyses show that nuclear and cytoplasmic RNA levels are differentially affected by dioxin". Biochim. Biophys. Acta. 1759 (8–9): 388–402. doi:10.1016/j.bbaexp.2006.07.005. PMID 16962184.
- Gilbert, Scott F.; Barresi, Michael J. F. (2003). Developmental Biology. Sinauer Associates. .
- Keene JD, Komisarow JM, Friedersdorf MB (2006). "RIP-Chip: the isolation and identification of mRNAs, microRNAs and protein components of ribonucleoprotein complexes from cell extracts". Nat Protoc. 1 (1): 302–7. doi:10.1038/nprot.2006.47. PMID 17406249. مؤرشف من الأصل في 28 يوليو 2019.