Chez les eucaryotes (organismes à noyau), l’épissage est un processus par lequel les ARN transcrits à partir de l'ADN génomique peuvent subir des étapes de coupure et ligature qui conduisent à l'élimination de certaines régions dans l’ARN final. Les segments conservés s’appellent des exons et ceux qui sont éliminés s’appellent des introns.
Les gènes sont donc constitués d’une suite d’exons et d’introns alternés ; ceci s’observe principalement dans les gènes codant des protéines, mais aussi dans certains gènes d’ARN non codants, comme ceux des ARNt.
Lors de la transcription de gènes codant des protéines, un ARN prémessager est synthétisé puis est épissé dans le noyau de la cellule pour donner lieu à l'ARN messager dit mature. L’ARNm mature, constitué des seuls exons, est alors exporté vers le cytoplasme pour être traduit en protéine.
Il a été démontré que des mécanismes de contrôle s'assurent que les ARNm ont été correctement épissés avant de permettre leur exportation[1].
L’épissage est catalysé par un ensemble de complexes ribonucléoprotéiques appelé collectivement splicéosome (épissage se disant splicing en anglais). Chaque complexe, appelé petite ribonucléoprotéine nucléaire, contient un ARN et plusieurs protéines. L'épissage des ARNm est également catalysé par les snARN (small nuclear ARN) qui sont de petits ARN non codants liés à des protéines.
Il existe également des introns appelés autoépissables ou autocatalytiques, c’est-à-dire capables de s’exciser sans intervention d’un splicéosome, dans les mitochondries, les plastes et certaines bactéries. Cependant, au moins dans les mitochondries et les chloroplastes, certains de ces introns nécessitent l’intervention de protéines nucléaires.
Le mécanisme catalytique du splicéosome est encore imparfaitement connu, mais par analogie avec le fonctionnement du ribosome, on pense que c'est l’ARN qui est catalytique (et donc que le splicéosome est un ribozyme).
L'épissage est beaucoup plus long que la transcription, cette dernière durant quelques minutes contre environ une heure et demie pour l'épissage.
Structure des introns
Les jonctions intron/exon contiennent des séquences nucléotidiques caractéristiques qui sont conservées. Ces séquences sont reconnues par le splicéosome. L'intron contient en plus une séquence interne, dite boîte de branchement. Cette boîte de branchement comporte une adénosine qui joue un rôle central dans le processus d'épissage.

L'épissage proprement dit s'effectue en deux temps, on a tout d'abord une attaque nucléophile du 2'-OH du ribose de l'adénosine de la boite de branchement sur le phosphate de la jonction exon-intron en 5'. Après cette coupure, le 3'-OH libéré au niveau de l'exon amont attaque le phosphate de la jonction intron-exon en aval. Les produits de cette réaction sont d'une part les deux exons ligaturés correctement et d'autre part, l'intron cyclisé au niveau de l'adénosine de la boîte de branchement. En raison de sa forme particulière, cette forme de l'intron est appelée lasso (en: lariat). Le lasso est finalement ouvert par une enzyme de débranchement afin de pouvoir être recyclé

Mécanisme d'épissage
Petites ribonucléoprotéines nucléaires
Les petites ribonucléoprotéines nucléaires (pRNPn ou snRNP) sont des composants du splicéosome présents dans le noyau des cellules eucaryotes. Le processus d'épissage canonique fait appel à cinq pRNPn, appelés U1, U2, U4, U5 et U6. Chacun de ces pRNPn est composé d'un ARN, appelé ARNpn, qui adopte une structure secondaire et tertiaire spécifique, et de plusieurs protéines. Parmi les protéines associées, certaines sont communes à tous les pRNPn et d'autres sont spécifiques de chacun d'entre eux. Les protéines communes sont appelées protéines Sm, elles sont au nombre de sept et s'associent pour former un anneau heptamérique entourant un segment du snARN.
Les protéines spécifiques sont en nombre variable, il y en a par exemple trois pour U1: U1A, U1C et U1-70K.
Processus d'épissage
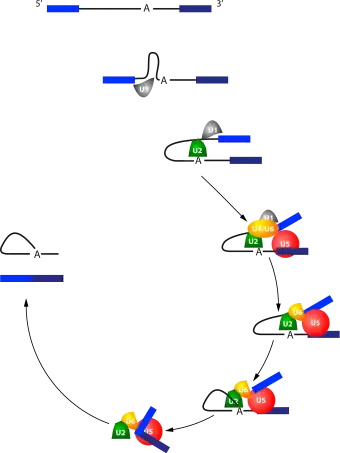
Le processus d'épissage est un mécanisme dynamique qui suit une succession précise d'évènements au cours de laquelle les différentes petites ribonucléoprotéines nucléaires s'assemblent et se désassemblent du splicéosome. La séquence est la suivante :
- U1 s'associe à la jonction 5' de l'intron ;
- U2 s'associe à la boite de branchement ;
- U4 associé à U6 rapproche U1 et U2, réalisant ainsi un pont entre la jonction 5' de l'intron et la boite de branchement ;
- U5 s'associe à son tour et rapproche les bords 3' et 5' des exons à suturer ;
- U4 et U1 quittent le complexe ;
- Le 2'-OH du A de la boite de branchement coupe la jonction 5' de l'intron ;
- Le 3'-OH du nucléotide en 3' de l'exon amont coupe l'autre jonction ;
- L'ARNm épissé et l'intron en « lasso » sont libérés.
Association des composants du splicéosome
La plupart des interactions entre l'ARNm et les petites ribonucléoprotéines nucléaires passent par des appariements de base entre portions de séquences nucléotidiques complémentaires. Ainsi par exemple le snARN U1 contient une séquence complémentaire de la séquence consensus retrouvée à la jonction exon-intron en 5', ce qui lui permet de se lier à cette région des pré-ARN messagers. Il existe également des appariements ARN-ARN entre les différents snARN.
Splicéosome mineur
On a découvert qu'un petit nombre d'introns atypiques était épissé par un second spliceosome, appelé spliceosome mineur. On appelle parfois ces introns des introns ATAC, car les premiers qui ont été découverts étaient bordés de part et d'autre par des séquences AT (en 5') et AC (en 3'), au lieu des séquences conservées GU (en 5') et AG (en 3') des introns classiques. Le splicéosome mineur fonctionne suivant un schéma proche du splicéosome classique, mais avec d'autres petites ribonucléoprotéines nucléaires : U11, U12, U4atac et U6atac qui remplacent U1, U2, U4 et U6. Le splicéosome mineur utilise en revanche U5, comme le splicéosome majeur.
Introns autocatalytiques
Chez plusieurs types d'organismes (protistes, champignons, plantes), certains ARN introniques n'ont pas besoin de splicéosome pour leur excision, ils sont capables d'assurer leur propre épissage de manière autonome (self-splicing introns), sans intervention des snRNP[2]. On les trouve en particulier fréquemment dans les ARN produits dans les organites (mitochondries et chloroplastes) : ARNm, ARNt et ARNr. Ces introns sont hautement structurés et sont dotés d'une activité catalytique intrinsèque, ce sont des ribozymes. Certains de ces introns sont entièrement autonomes et d'autres requièrent l'intervention de protéines accessoires.
On a identifié deux grandes familles d'introns autocatalytiques, sur la base de leur structure 3D : les introns de groupes I et les introns de groupe II. Les introns de groupe II présentent des ressemblances structurales avec les ARN des snRNP du splicéosome et on pense qu'ils pourraient dériver de la même origine[3]. Les introns de groupe II catalysent par exemple une excision produisant une structure en lasso, comme c'est le cas avec le splicéosome classique : les introns du GII se replient pour réaliser deux estérifications. On obtient donc d'un côté l'intron clivé et de l'autre les exons soudés.
Épissage alternatif
L'épissage alternatif fut observé pour la première fois en 1977 et constitue l’un des mécanismes les plus complexes chez les eucaryotes[4]. Les chercheurs ont découvert que le premier ARN transcrit produit par le virus adénovirus type 2 dans sa phase tardive subissait des voies d'épissage différentes, donnant naissance à des mARNs codant différentes protéines virales. C'est en 1981, que le premier exemple d'un transcrit d'épissage alternatif fut caractérisé, d'un gène endogène « normal »[5]. L'équipe de chercheurs a découvert que le gène codant la calcitonine, une hormone thyroïdienne, subissait de l'épissage alternatif dans les cellules mammifères. Depuis, l'épissage alternatif est admis comme étant omniprésent dans les cellules de mammifères[6].
Le splicéosome reconnaît des signaux d'épissage, comme pour un signal radio, ces signaux d'épissage sont plus ou moins forts, ce qui implique que le splicéosome les reconnait plus ou moins bien.
Ces signaux sont simplement des séquences spécifiques de nucléotides.
Les signaux faibles sont appelés « signaux d'épissage alternatif », ils vont permettre à un pre-ARNm d'être épissé en plusieurs ARNm matures. Par opposition les signaux forts sont appelés « signaux constitutifs ».
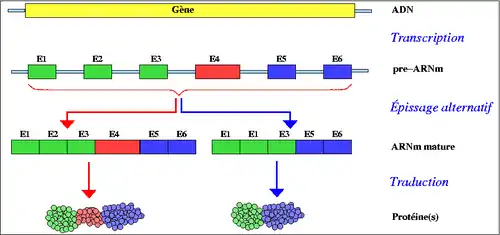
C'est ainsi qu'un gène peut coder plusieurs protéines. L'épissage alternatif joue un rôle très important dans le développement des cellules, l'organisation des tissus et même dans le développement d'un individu. (par exemple : le gène Slx pour la différenciation du sexe chez la drosophile). « On pense qu'au moins 70 % des quelque 30 000 gènes qui composent le génome humain subissent un épissage alternatif et que, en moyenne, un gène donne naissance à 4 variants issus d'un tel épissage, pouvant donner naissance à environ 100 000 protéines différentes de par leur séquence et, du coup, leurs activités[7] - [8]. » Le dogme « 1 gène pour 1 protéine » n'est plus généralement valable et « 1 gène, des protéines » correspond plus à la réalité.
Dans certains cas extrêmes, l'épissage alternatif permet à un seul gène de coder plus de protéines que tous les autres réunis. C'est le cas pour Dscam chez la drosophile qui peut coder jusqu'à 38016 ARN messagers différents[9].
Cinq modes d’épissage alternatif sont généralement reconnus soit[6],[10] :
- Exon cassette (Cassette exons) : dans ce cas, les exons peuvent subir l’épissage du transcrit primaire ou être retenus dans celui-ci. C’est le mode d’épissage alternatif le plus courant dans les cellules de mammifères ;
- Exons mutuellement exclusifs (mutually exclusive exon) : un des deux exons est retenu ou exclu dans le mARN après l’épissage, mais pas les deux à la fois ou aucun ;
- Extrémité 5` alternative (Alternative 5` splice site) : généré par les multiples sites de promoteurs ;
- Extrémité 3` alternative (Alternative 3` splice site) : généré par les multiples sites de polyadénylations ;
- La rétention d’introns (Intron rétention) : une séquence peut être excisée ou retenue d’un intron. Si l’intron retenu est dans la région codante, l’intron pourra encoder des acides aminés dans le cadre de lecture des exons adjacent, ou coder un codon STOP, ou causer un saut dans le cadre de lecture menant à des protéines tronquées. Ce mode est rarement observé dans les cellules de mammifère.
En plus des cinq modes énumérés précédemment, les événements suivants permettent de produire une grande variété de protéines alternatives :
- Le lasso ne comprend pas toujours un intron seul. Pour un gène contenant un grand nombre d'exons, le lasso formé peut comprendre 2 introns et 1 exon au lieu d'un seul intron. On aura alors des exons non-représentés dans l'ARNm mature, et donc dans la protéine ;
- On peut trouver plusieurs promoteurs au sein d'un même gène. De ce fait, les ARNm matures ne contiennent que les exons en aval du promoteur, ce qui entraine la traduction de plusieurs protéines différentes selon le promoteur utilisé lors de l'épissage ;
- On peut trouver des séquences polyA (séquence de résidus adénine qui protège l'ARNm des exonucléases) à l'intérieur de l'ARNm en plus de la séquence polyA que l'on trouve à l'extrémité 3' de l'ARNm. Selon la séquence polyA qui sera coupée avant traduction, on aura donc des protéines traduites différentes.
Il a été proposé que chez les eucaryotes, l’épissage alternatif est une étape très importante pour une plus grande efficacité, car l’information peut être «stockée» plus économiquement. Plusieurs protéines peuvent être codées par un seul gène, c’est ce qui mène au protéome alternatif[6]. Le protéome alternatif est l’ensemble des protéines alternatives produites par un organisme et fait partie du protéome. L’étude de l’épissage alternatif à l’échelle d’un génome est un des challenges actuels que rencontre la biologie moléculaire. Avec l’avènement des techniques de haute résolution il devient de plus en plus facile d’étudier les évènements d’épissage alternatif. En voici quelques exemples[11] :
- Puce à exons : dans les cas des puces à exons, le but n’est pas de détecter l’expression génétique mais les différentes formes d’épissages présents dans les cellules. La puce contient des sondes couvrant toutes les formes d’épissages alternatifs connus pour les ARN ;
- Séquençage de l’ARN : le séquençage de l’ARN (RNAseq) est une technique récemment développée qui utilise les technologies nouvelle génération du séquençage de l’ADN pour étudier le transcriptome ;
- iCLIP (individual-nucleotide Resolution UV Cross-linking and Immunoprecipitation) : Est une méthode de biologie moléculaire qui permet de détecter les intéractions protéine/ARN in vivo et de déterminer la séquence exacte reconnue par la protéine. Cette méthode utilise la photoréactivité, une propriété naturelle qui s’applique aux nucléotides permettant la liaison de l’ARN et de l’ADN.
De plus, il est aussi possible d’analyser les résultats obtenus de la biologie moléculaire par bio-informatique réduisant grandement le temps d’analyse requis. Des logiciels tels que : Genome Browser, ARN splicing map et Heatmap.
L'épissage alternatif est un processus important de régulation de l'expression des gènes. Deux conséquences de l'épissage alternatif y concourent :
La première est connue sous les initiales NMD Nonsense mediated decay : lors de l'épissage alternatif, l'ajout ou le retrait d'exon, ou bien encore la modification de la longueur de tel ou tel exon (par rapport à l'ARNm « canonique ») peut entraîner un décalage du cadre de lecture (les nucléotides sont lus trois par trois par le ribosome lors de la traduction), et de ce fait causer l'apparition prématurée d'un codon STOP, ce qu'on appelle une mutation non-sens. Les ARNm ainsi formés sont reconnus par un ensemble de protéines et dégradés avant que la traduction puisse s'opérer. Ce processus n'est pas seulement un mécanisme de « contrôle qualité » mais permet aussi, en augmentant le taux des épissages alternatifs producteurs de codons STOP prématurés, de diminuer le nombre de protéines traduites. La seconde conséquence est tout simplement le fait qu'un changement dans la séquence de l'ARNm se répercute sur celle de la protéine et donc potentiellement sur ses capacités physico-chimiques, et permet de réguler sa fonction. Par exemple : la protéine CD45 joue un rôle dans l'activation des lymphocytes T lors d'une réponse immunitaire à une infection. Lorsque les lymphocytes T sont au repos et donc potentiellement activables, la forme la plus longue de la protéine est exprimée (elle comporte la totalité des neuf exons). Lorsque les lymphocytes T viennent d'être activés, les ARNm de CD45 sont épissés alternativement et les exons 4,5,6 sont excisés. La forme de CD45 ainsi produite est donc plus courte et ne peut pas jouer son rôle d'activateur. Ceci empêche les lymphocytes T de recevoir un signal d'activation trop longtemps et donc de déclencher une réponse immunitaire disproportionnée.
Un épissage alternatif anormal a aussi été trouvé dans une large proportion de cellules cancéreuses. Jusqu’à présent, il n’était pas défini quel rôle un épissage alternatif anormal intervenait sur la croissance des cellules. Il a été démontré que c’est une réduction de l’épissage alternatif dans les cellules cancéreuses, comparé à des cellules normales, qui causerait certains cancers. De plus, les cellules cancéreuses auraient plus de rétention d’introns que les cellules normales, mais moins d’exon cassette[12].
Épissage et ARN circulaires
L'existence de l’épissage en arrière ou rétro-épissage (backsplicing en anglais) fut suggérée la première fois en 2012[13]. Cet épissage en arrière permet d’expliquer la genèse des ARN circulaires résultant de l’exacte jonction entre l’extrémité 3’ d’un exon avec l’extrémité 5' d’un (autre) exon situé en amont[14]. Dans ces ARN circulaires, dits exoniques, la jonction est une liaison 3’-5’ classique.
L’exclusion des séquences introniques lors de l’épissage peut aussi laisser des traces, sous la forme d’ARN circulaires[15]. En effet, dans certains cas, la partie circulaire du lasso n’est pas détruite et forme un ARN circulaire[16]. La jonction de circularisation est une liaison 2'-5'.
Notes et références
- ↑ (en) Daniel Zenklusen et Françoise Stutz, « Nuclear export of mRNA », FEBS Letters, vol. 498, , p. 150–156 (ISSN 1873-3468, DOI 10.1016/S0014-5793(01)02482-6, lire en ligne, consulté le )
- ↑ T R Cech, « Self-Splicing of Group I Introns », Annual Review of Biochemistry, vol. 59, , p. 543–568 (ISSN 0066-4154, DOI 10.1146/annurev.bi.59.070190.002551, lire en ligne, consulté le )
- ↑ (en) Mahadevan Seetharaman, Nadukkudy V. Eldho, Richard A. Padgett et Kwaku T. Dayie, « Structure of a self-splicing group II intron catalytic effector domain 5: Parallels with spliceosomal U6 RNA », RNA, vol. 12, , p. 235–247 (ISSN 1355-8382 et 1469-9001, PMID 16428604, PMCID 1370903, DOI 10.1261/rna.2237806, lire en ligne, consulté le )
- ↑ Chow LT, Gelinas RE, Broker TR, Roberts RJ (1977). « An amazing sequence arrangement at the 5' ends of adenovirus 2 messenger RNA ». Cell 12 (1) : 1–8.
- ↑ Leff SE, Rosenfeld MG, Evans RM (1986). « Complex Transcriptional Units: Diversity in Gene Expression by Alternative RNA Processing ». Annual Review of Biochemistry (en) 55 (1) : 1091–117.
- 1 2 3 Black, Douglas L. (2003). « Mechanisms of alternative pre-messenger RNA splicing ». Annual Review of Biochemistry (en) 72 (1) : 291–336.
- ↑ Un article sur le net. Epissage alternatif. (2009). ExonHit Therapeutics.
- ↑ Hewetson A, Wright-Pastusek AE, Helmer RA, Wesley KA, Chilton BS: Conservation of inter-protein sites in RUSH and RFBP, an ATP11B isoform. Mol Cell Endocrinol. (2008) Sep 24;292(1-2):79-86.
- ↑ AM Celotto, BR Graveley, Genetics, Vol. 159, No. 2. (October 2001), pp. 599-608.
- ↑ Pan, Q; Shai O, Lee LJ, Frey BJ, Blencowe BJ (Dec 2008). « Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing ». Nature Genetics 40 (12) : 1413–1415.
- ↑ Alberts, B. et al., Molecular Biology of the Cell in Cell 5th, vol. 54, États-Unis, 2008, 5e édition.
- ↑ Kim E, Goren A, Ast G (2008). « Insights into the connection between cancer and alternative splicing ». Trends in Genetics 24 (1) : 7–10.
- ↑ Salzman J, Gawad C, Wang PL, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types. PLoS One 2012;7(2):e30733.
- ↑ Jeck WR, Sorrentino JA, Wang K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats. RNA 2013;19(2):141-57.
- ↑ Zhang Y, Zhang XO, Chen T, et al. Circular intronic long noncoding RNAs. Molecular cell 2013;51(6):792-806.
- ↑ Talhouarne GJ and Gall JG. Lariat intronic RNAs in the cytoplasm of Xenopus tropicalis oocytes. RNA 2014;20(9):1476-87.
Voir aussi
Articles connexes
- Épissure