يشير علم الحفريات الجزيئيإلى استعادة وتحليل الحمض النووي والبروتينات والكربوهيدرات أو الدهون ومنتجاتها الديوجينية من بقايا الإنسان والحيوان والنبات القديمة. وقد أثمر مجال علم الحفريات الجزيئية رؤى هامة في الأحداث التطورية، وشتات الأنواع، واكتشاف وتوصيف الأنواع المنقرضة. من خلال تطبيق التقنيات التحليلية الجزيئية على الحمض النووي في الحفريات، يمكن للمرء أن يحدد مستوى الصلة بين أي كائنين تم استرداد الحمض النووي من هما.
وقد سمحت التطورات في مجال علم الحفريات الجزيئية للعلماء بمتابعة أسئلة تطورية على المستوى الوراثي بدلاً من الاعتماد على الاختلاف البذيبي وحده. باستخدام تقنيات التكنولوجيا الحيوية المختلفة مثل عزل الحمض النووي، والتضخيم، وتسلسل العلماء تمكنت من الحصول على رؤى جديدة موسعة في الاختلاف والتاريخ التطوري لعدد لا يحصى من الكائنات الحية.
التاريخ
ويقال إن دراسة علم الحفريات الجزيئية قد بدأت مع اكتشاف أبيلسون للأحماض الأمينية 360 مليون سنة المحفوظة في قذائف الأحفورية. ومع ذلك، فإن Svante Pääbo هو في كثير من الأحيان واحد يعتبر مؤسس مجال علم الحفريات الجزيئية.
وقد شهد مجال علم الحفريات الجزيئية عدة تطورات رئيسية منذ الخمسينات وهو مجال ينمو باستمرار. وفيما يلي جدول زمني يبين المساهمات البارزة التي تم تقديمها.
الجدول الزمني
منتصف الخمسينيات: وجد أبيلسون أحماض أمينية محفوظة في قذائف أحفورية عمرها حوالي 360 مليون سنة. فكرة منتجة لمقارنة تسلسلات الأحماض الأمينية الأحفورية بالكائن الموجود حتى يمكن دراسة التطور الجزيئي. [1]
1970s: يتم دراسة الببتيدات الأحفورية عن طريق تحليل الأحماض الأمينية . [2] ابدأ في استخدام الببتيدات الكاملة . [3]
أواخر السبعينيات: درس علماء النبات الحفريات (يمكن تهجئتها أيضًا باسم علماء الحفريات القديمة ) الجزيئات من النباتات الأحفورية المحفوظة جيدًا. [4]
1984: أول تسلسل ناجح للحمض النووي لفصيلة منقرضة، وهو quagga ، وهو نوع شبيه بالحمار الوحشي. [5]
1991: مقالة منشورة عن الاستخراج الناجح للبروتينات من العظام الأحفورية للديناصورات، وتحديداً الزلازل. [6]
2005: إحياء العلماء لفيروس إنفلونزا 1918 المنقرض. [7]
2006: بدأ تحليل ونشر مقاطع تسلسل الحمض النووي للنياندرتال . [8]
2007: قام العلماء بتجميع فيروس رجعي داخلي منقرض كامل (HERV-K) من الصفر. [9]
2010: اكتشاف نوع جديد من البشر في وقت مبكر، Denisovans ، من جينوم الميتوكوندريا والنووية المستعادة من العظام الموجودة في كهف في سيبيريا. أظهر التحليل أن عينة دينيسوفان عاشت منذ 41000 عام تقريبًا، وشاركت سلفًا مشتركًا مع كل من البشر الحديثين ونياندرتال قبل حوالي مليون سنة في أفريقيا. [10]
2013: تم بنجاح تسلسل أول جينوم نياندرتال كامل. يمكن العثور على مزيد من المعلومات في مشروع جينوم نياندرتال . [11]
2013: تسلسل عينة عمرها 400000 عام مع الحمض النووي المتبقي المتسلسل وتبين أنها سلف مشترك لنياندرتال ودينيسوفانز، سميت فيما بعد Homo heidelbergensis . [12]
عام 2015: تم الإبلاغ عن سن أحفوري عمره 110,000 عام يحتوي على DNA من Denisovans . [13] [14]
كواجا
كان أول تسلسل ناجح للحمض النووي لأنواع منقرضة في عام 1984 ، من عينة متحف عمرها 150 عامًا من quagga ، وهو نوع يشبه الحمار الوحشي. [5] الحمض النووي (المعروف أيضا باسم الحمض النووي الميتوكوندري) كان التسلسل من المجفف عضلة كواجا، ووجد أن تختلف بنسبة 12 التبديلات الأساسية من الحمض النووي من الحمار الوحشي الجبلية. استنتج أن هذين النوعين كان لهما سلف مشترك قبل 3-4 مليون سنة، وهو ما يتوافق مع الأدلة الأحفورية المعروفة للأنواع. [15]
دينيسوفانز
تم اكتشاف Denisovans of Eurasia ، وهو نوع من البشر له علاقة بالإنسان البدني والبشر، كنتيجة مباشرة لتسلسل الحمض النووي لعينة عمرها 41000 عام تم استردادها في عام 2008. أظهر تحليل الحمض النووي للميتوكوندريا من عظمة إصبع تم استردادها أن العينة متميزة وراثيًا عن كل من البشر والنياندرتال. تم العثور على سنين وعظم إصبع القدم لاحقًا ينتمون إلى أفراد مختلفين من نفس السكان. يشير التحليل إلى أن كلا من إنسان نياندرتال ودينيسوفان كانوا حاضرين بالفعل في جميع أنحاء أوراسيا عندما وصل البشر الحديثون. [11] في نوفمبر 2015 ، أفاد العلماء عن العثور على سن أحفوري يحتوي على الحمض النووي من دنيسوفانز ، ويقدر عمره بـ 110,000 سنة. [13] [14]
تحليل الحمض النووي للميتوكوندريا

يختلف mtDNA من عظمة إصبع Denisovan عن مثيله لدى البشر المعاصرين بـ 385 قاعدة ( نيوكليوتيدات ) في حبلا mtDNA من حوالي 16500 ، في حين أن الفرق بين البشر الحديثين ونياندرتال حوالي 202 قاعدة. في المقابل، يبلغ الفرق بين الشمبانزي والبشر الحديثين حوالي 1,462 زوجًا من الحمض النووي الميتوكوندري. [10] يشير هذا إلى وقت اختلاف منذ حوالي مليون سنة. يحمل الحمض النووي الميتوكوندري من السن تشابهًا كبيرًا مع عظم الإصبع، مما يشير إلى أنها تنتمي إلى نفس السكان. [16] من السن الثانية، تم استعادة تسلسل mtDNA الذي أظهر عددًا كبيرًا بشكل غير متوقع من الاختلافات الجينية مقارنة بتلك الموجودة في الأسنان الأخرى والإصبع، مما يشير إلى درجة عالية من تنوع mtDNA. أظهر هذان الشخصان من نفس الكهف تنوعًا أكثر مما شوهد بين عينات النياندرتال من جميع أنحاء أوراسيا، وكانوا مختلفين مثل البشر في العصر الحديث من قارات مختلفة. [17]
تحليل الجينوم النووي
كما تم عزل وتسلسل DNA النووي من عظم إصبع Denisova. أظهرت هذه العينة درجة غير عادية من الحفاظ على الحمض النووي ومستوى منخفض من التلوث. كانوا قادرين على تحقيق تسلسل جينومي شبه كامل، مما سمح بإجراء مقارنة تفصيلية مع البشر البدائيين والإنسان الحديث. من هذا التحليل، استنتجوا، على الرغم من الاختلاف الواضح في تسلسل الميتوكوندريا لديهم، تقاسم سكان دينيسوفا مع نياندرتال فرعًا مشتركًا من النسب المؤدي إلى البشر الأفريقيين الحديثين. إن متوسط وقت الاختلاف المقدر بين تسلسل دينيسوفان ونياندرتال هو 640,000 سنة مضت، والوقت بين هذين التسلسلين وتسلسل الأفارقة المعاصرين هو 804,000 سنة مضت. يقترحون أن الاختلاف في Denisova mtDNA ينتج إما من استمرار سلالة تم تطهيرها من الفروع الأخرى للبشرية من خلال الانجراف الوراثي أو من التدخل من سلالة هومين أقدم. [16]
هومو هايدلبرغينس

تم اكتشاف Homo heidelbergensis لأول مرة في عام 1907 بالقرب من هايدلبرغ، ألمانيا، ثم تم العثور عليه أيضًا في مكان آخر في أوروبا وأفريقيا وآسيا. [18] [19] ومع ذلك، لم يتم العثور حتى عام 2013 على عينة ذات حمض نووي قابل للاسترجاع، في عظم الفخذ الذي يبلغ عمره حوالي 400000 عام والذي تم العثور عليه في كهف سيما دي لوس هويسوس في إسبانيا. تم العثور على عظم الفخذ يحتوي على كل من الحمض النووي الميتوكوندري والحمض النووي. سمحت التحسينات في استخلاص الحمض النووي وتقنيات تحضير المكتبة لعزل mtDNA وتسلسله بنجاح، ولكن تم العثور على الحمض النووي المتحلل في العينة المرصودة، وكان ملوثًا أيضًا بالحمض النووي من دب كهف قديم ( Ursus deningeri ) موجود في الكهف. وجد تحليل mtDNA رابطًا مدهشًا بين العينة و Denisovans ، وأثارت هذه النتيجة العديد من الأسئلة. تم اقتراح العديد من السيناريوهات في ورقة يناير 2014 بعنوان "تسلسل جينوم الميتوكوندريا ل Hominin من Sima de los Huesos" ، مما يوضح عدم التقارب في المجتمع العلمي حول كيفية ارتباط Homo heidelbergensis بمجموعات hominin المعروفة الأخرى. أحد السيناريو المعقول الذي اقترحه المؤلفون هو أن H. heidelbergensis كان سلفًا لكل من Denisovans و Neanderthals. [20] تشير الجينومات النووية المتسلسلة بالكامل من كل من Denisovans و Neanderthals إلى سلف مشترك منذ ما يقرب من 700000 سنة، ويقترح أحد الباحثين البارزين في المجال، Svante Paabo ، أنه ربما تكون هذه المجموعة البشرية الجديدة هي تلك السلف المبكر. [12]
التطبيقات
ساهمت تقنيات علم الحفريات الجزيئية المطبقة على الحفريات في اكتشاف وتوصيف العديد من الأنواع الجديدة، بما في ذلك Denisovans و Homo heidelbergensis . لقد تمكنا من فهم أفضل للطريق الذي سلكه البشر وهم يسكنون الأرض، وما هي الأنواع التي كانت موجودة خلال هذا الشتات .
الإنقراض
من الممكن الآن إحياء الأنواع المنقرضة باستخدام تقنيات علم الحفريات الجزيئية. تم تحقيق ذلك لأول مرة عن طريق الاستنساخ في عام 2003 مع Pyrenean ibex ، وهو نوع من الماعز البري الذي انقرض في عام 2000. تم حقن نوى من خلايا Pyrenean ibex في بيوض الماعز التي تم تفريغها من الحمض النووي الخاص بها، وزرعت في أمهات الماعز البديلة. [21] عاش النسل بعد سبع دقائق فقط من الولادة، بسبب عيوب في رئتيه. وقد لوحظ أن الحيوانات المستنسخة الأخرى لديها عيوب رئوية مماثلة. [22]
هناك العديد من الأنواع التي انقرضت كنتيجة مباشرة للنشاط البشري. وتشمل بعض الأمثلة على طائر الدودو ، و أوك عظيم ، و نمر تسمانيا ، و الدلفين النهري الصيني ، و الحمام الزاجل . يمكن إحياء الأنواع المنقرضة باستخدام استبدال الأليلات [23] من الأنواع وثيقة الصلة التي لا تزال حية. من خلال الحاجة فقط إلى استبدال عدد قليل من الجينات داخل الكائن الحي، بدلاً من الاضطرار إلى بناء جينوم الأنواع المنقرضة من الصفر، يمكن إعادة العديد من الأنواع بهذه الطريقة، حتى إنسان نياندرتال. الأخلاق المحيطة بإعادة إدخال الأنواع المنقرضة مثيرة للجدل للغاية. يزعم منتقدو إعادة الأنواع المنقرضة إلى الحياة أنها ستحول الأموال والموارد المحدودة من حماية مشاكل التنوع البيولوجي الحالية في العالم. [24] مع اقتراب معدلات الانقراض الحالية من 100 إلى 1000 ضعف معدل الانقراض في الخلفية، [25] يخشى أن يقلل برنامج الانقراض من مخاوف الجمهور بشأن أزمة الانقراض الجماعي الحالية، إذا كان يعتقد أنه يمكن جلب هذه الأنواع ببساطة العودة إلى الحياة. كما يطرح محررو مقال علمي أمريكي عن إزالة الانقراض: هل علينا إعادة الماموث الصوفي فقط للسماح للأفيال بالانقراض في هذه الأثناء؟ العامل الرئيسي الدافع لانقراض معظم الأنواع في هذه الحقبة (بعد 10000 سنة قبل الميلاد) هو فقدان الموائل، ولن تؤدي إعادة الأنواع المنقرضة مؤقتًا إلى إعادة خلق البيئة التي كانت تعيش فيها. [26]
يتحدث أنصار إزالة الانقراض، مثل كنيسة جورج ، عن العديد من الفوائد المحتملة. يمكن أن يساعد إعادة إدخال أنواع حجر الزاوية المنقرضة، مثل الماموث الصوفي ، في إعادة التوازن بين النظم البيئية التي كانت تعتمد عليها في السابق. يمكن لبعض الأنواع المنقرضة أن تخلق فوائد واسعة للبيئات التي كانت تسكنها ذات مرة، إذا عادت. على سبيل المثال، قد يكون الماموث الصوفي قادرًا على إبطاء ذوبان التندرا الروسية والقطبية الشمالية بعدة طرق مثل تناول العشب الميت بحيث يمكن للعشب الجديد أن ينمو ويتجذر، ويكسر الثلج دوريًا، مما يعرض الأرض أسفل القطب الشمالي الهواء. يمكن أيضًا استخدام هذه التقنيات لإعادة إدخال التنوع الجيني في الأنواع المهددة، أو حتى إدخال جينات وسمات جديدة للسماح للحيوانات بالتنافس بشكل أفضل في بيئة متغيرة. [27]
البحث والتكنولوجيا
عندما يتم العثور على عينة محتملة جديدة، يقوم العلماء عادةً بتحليل أولاً للحفاظ على الخلايا والأنسجة باستخدام التقنيات النسيجية ، واختبار شروط بقاء الحمض النووي. سيحاولون بعد ذلك عزل عينة من الحمض النووي باستخدام التقنية الموضحة أدناه، وإجراء تضخيم PCR للحمض النووي لزيادة كمية الحمض النووي المتاحة للاختبار. ثم يتم تسلسل هذا الحمض النووي المكبر. يتم الحرص على التحقق من أن التسلسل يتطابق مع السمات الوراثية للسلالة للكائن الحي. [5] عندما يموت كائن حي، يمكن استخدام تقنية تسمى تأريخ الأحماض الأمينية لتقدم عمر الكائن الحي. أن يتفقد درجة تروسم من حمض الأسبارتيك ، ليسين ، و ألانين داخل الأنسجة. مع مرور الوقت، تزداد نسبة D / L (حيث "D" و "L" هي صور معكوسة لبعضها البعض) من 0 إلى 1. [28] في العينات التي تكون فيها نسبة D / L لحمض الأسبارتيك أكبر من 0.08 ، لا يمكن استرداد تسلسلات DNA القديمة (اعتبارًا من عام 1996). [29]
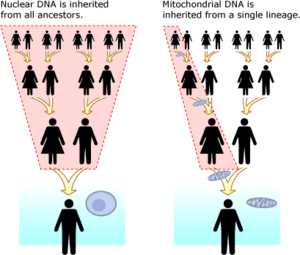
الميتوكوندريا DNA مقابل DNA النووي
الميتوكوندريا DNA (mtDNA) منفصلة عن الحمض النووي النووي. وهي موجودة في عضيات تسمى الميتوكوندريا في كل خلية . على عكس الحمض النووي النووي ، الموروث من كلا الوالدين وإعادة ترتيبه كل جيل، تنتقل نسخة دقيقة من الحمض النووي من الميتوكوندريا من الأم إلى أبنائها وبناتها. فوائد إجراء تحليل الحمض النووي مع الحمض النووي للميتوكوندريا هو أنه يحتوي على معدل طفرة أصغر بكثير من الحمض النووي النووي، مما يجعل تتبع الأنساب على مقياس عشرات الآلاف من السنين أسهل بكثير. بمعرفة معدل الطفرة الأساسي لـ mtDNA ، [30] (يُعرف هذا المعدل في البشر أيضًا باسم الساعة الجزيئية للميتوكوندريا البشرية ) يمكن للمرء تحديد مقدار الوقت الذي تم فيه فصل أي خطين. ميزة أخرى لـ mtDNA هي وجود آلاف النسخ منه في كل خلية، في حين توجد نسختان فقط من الحمض النووي النووي في كل خلية. [31] جميع حقيقيات النوى ، وهي مجموعة تضم جميع النباتات والحيوانات والفطريات، تحتوي على mtDNA. [32] عيب mtDNA هو أنه يتم تمثيل خط الأم فقط. على سبيل المثال، سيرث الطفل 1/8 من الحمض النووي الخاص به من كل من أجداده الثمانية، إلا أنه سيرث استنساخًا دقيقًا لـ mtDNA لجدة جدته الأم. هذا مماثل لطفل يرث فقط الاسم الأخير لوالد جده الأبوي، وليس مزيجًا من جميع الألقاب الثمانية.
عزل
هناك العديد من الأشياء التي يجب مراعاتها عند عزل مادة. أولاً، اعتمادًا على ماهيتها ومكانها، هناك بروتوكولات يجب تنفيذها لتجنب تلوث العينة وزيادة تدهورها. [33] بعد ذلك، يتم التعامل مع المواد عادة في منطقة عمل معزولة ماديًا وتحت ظروف محددة (مثل درجة حرارة محددة، رطوبة، إلخ ...) أيضًا لتجنب التلوث وفقدان إضافي للعينة.
بمجرد الحصول على المادة، اعتمادًا على ماهيتها، هناك طرق مختلفة لعزلها وتنقيتها. يعد استخراج الحمض النووي من الأحافير أحد الممارسات الأكثر شيوعًا وهناك خطوات مختلفة يمكن اتخاذها للحصول على العينة المطلوبة. [33] يمكن أخذ الحمض النووي المستخرج من الأحافير المدفونة بالعنبر من عينات صغيرة وخلطه بمواد مختلفة، بالطرد المركزي ، والحضانة، والطرد المركزي مرة أخرى. من ناحية أخرى، يمكن إجراء استخراج الحمض النووي من الحشرات عن طريق طحن العينة، وخلطها مع العازلة، والتطهير من خلال أعمدة الألياف الزجاجية. [34] في النهاية، بغض النظر عن كيفية عزل العينة لهذه الأحافير، يجب أن يكون الحمض النووي المعزول قادرًا على التضخيم . [35]
تضخيم
استفاد مجال علم الحفريات الجزيئية بشكل كبير من اختراع تفاعل سلسلة البلمرة (PCR) ، والذي يسمح للمرء بعمل مليارات نسخ من جزء من DNA من نسخة واحدة محفوظة من DNA. كان أحد أكبر التحديات حتى هذه المرحلة هو الندرة الشديدة في الحمض النووي المستعاد بسبب تدهور الحمض النووي بمرور الوقت. [5]
التسلسل
يتم إجراء تسلسل الحمض النووي لتحديد ترتيب النيوكليوتيدات والجينات. [36] هناك العديد من المواد المختلفة التي يمكن استخراج الحمض النووي منها. في الحيوانات، يمكن استخدام كروموسوم الميتوكوندريا للدراسة الجزيئية. يمكن دراسة البلاستيدات الخضراء في النباتات كمصدر أساسي لبيانات التسلسل.
في النهاية، تُستخدم التسلسلات المتولدة لبناء أشجار تطورية . [36] تتضمن طرق مطابقة مجموعات البيانات: الحد الأقصى من الاحتمالية، والحد الأدنى من التطور (المعروف أيضًا باسم الانضمام إلى الجوار ) الذي يبحث عن الشجرة ذات أقصر طول إجمالي، وطريقة البخل القصوى التي تجد الشجرة التي تتطلب أقل عدد من التغييرات في حالة الأحرف. يمكن أيضًا تقييم مجموعات الأنواع المحددة داخل الشجرة في وقت لاحق عن طريق الاختبارات الإحصائية، مثل طريقة التمهيد، لمعرفة ما إذا كانت مهمة بالفعل.
القيود والتحديات
من الصعب الحصول على الظروف البيئية المثالية للحفاظ على الحمض النووي حيث تم تجفيف الكائن الحي وكشفه، بالإضافة إلى الحفاظ على حالته حتى التحليل. يتحلل الحمض النووي النووي عادة بسرعة بعد الموت بسبب عمليات التحلل المائي الداخلية ، [29] عن طريق الأشعة فوق البنفسجية، [5] والضغوط البيئية الأخرى.
أيضا، تم العثور على تفاعلات مع منتجات التحلل العضوي للتربة المحيطة للمساعدة في الحفاظ على المواد الجزيئية الحيوية. [37] ومع ذلك، فقد خلقت أيضًا التحدي الإضافي المتمثل في القدرة على فصل المكونات المختلفة حتى تتمكن من إجراء التحليل المناسب عليها. [38] كما تم العثور على بعض هذه الأعطال تتداخل مع عمل بعض الإنزيمات المستخدمة أثناء تفاعل البوليميراز المتسلسل.
أخيرًا، يعد التلوث أثناء تفاعل البوليميراز المتسلسل أحد أكبر التحديات في استخراج الحمض النووي القديم، خاصةً في الحمض النووي البشري القديم. يمكن أن تلوث كميات صغيرة من الحمض النووي البشري الكواشف المستخدمة لاستخراج و PCR الحمض النووي القديم. يمكن التغلب على هذه المشاكل من خلال العناية الصارمة في التعامل مع جميع الحلول وكذلك الأواني الزجاجية والأدوات الأخرى المستخدمة في العملية. يمكن أن يساعد أيضًا إذا قام شخص واحد فقط بإجراء عمليات الاستخلاص، لتقليل الأنواع المختلفة من الحمض النووي الموجودة. [29]
مقالات ذات صلة
المراجع
- Abelson, PH (1954). "Organic constituents of fossils". Carnegie Institute of Washington Yearbook. 53: 97–101.
- de Jong, EW; Westbroek P; Westbroek JF; Bruining JW (1974). "Preservation of antigenic properties of macromolecules over 70 Myr". Nature. 252 (5478): 63–64. doi:10.1038/252063a0. PMID 4139661.
- Westbrock P, van der Meide PH; van der Wey-Kloppers JS; et al. (1979). "Fossil macromolecules from cephalopod shells: Characterization, immunological response and diagenesis". Paleobiology. 5 (2): 151–167. doi:10.1017/S0094837300006448.
- Nitecki MH (1982). Biochemical Aspects of Evolutionary Biology. Chicago: University of Chicago Press. صفحات 29–91.
- Marota, Isolina; Franco Rollo (2002). "Molecular paleontology". Cellular and Molecular Life Sciences. 59 (1): 97–111. doi:10.1007/s00018-002-8408-8. PMID 11846037.
- Gurley, L. R; J. G. Valdez; W. D. Spall; B. F Smith; D. D. Gillette (February 1991). "Proteins in the fossil bone of the dinosaur, seismosaurus". Journal of Protein Chemistry. 10 (1): 75–90. doi:10.1007/BF01024658. PMID 2054066.
- Kaiser, J. (7 October 2005). "VIROLOGY: Resurrected Influenza Virus Yields Secrets of Deadly 1918 Pandemic" ( كتاب إلكتروني PDF ). Science. 310 (5745): 28–29. doi:10.1126/science.310.5745.28. PMID 16210501. مؤرشف من الأصل ( كتاب إلكتروني PDF ) في 4 مارس 201609 أبريل 2014.
- Dalton, Rex (15 May 2006). "Neanderthal DNA yields to genome foray". Nature. 441 (7091): 260–1. doi:. PMID 16710377.
- Rockefeller University (1 March 2007). "Ancient Retrovirus Is Resurrected". Science Daily. مؤرشف من الأصل في 1 فبراير 201609 أبريل 2014.
- Krause, Johannes; Fu, Qiaomei; Good, Jeffrey M.; Viola, Bence; Shunkov, Michael V.; Derevianko, Anatoli P.; Pääbo, Svante (24 March 2010). "The complete mitochondrial DNA genome of an unknown hominin from southern Siberia". Nature. 464 (7290): 894–897. Bibcode:2010Natur.464..894K. doi:. PMID 20336068.
- Prüfer K, Racimo F, Patterson N, Jay F, Sankararaman S, Sawyer S, Heinze A, Renaud G, Sudmant PH, de Filippo C, Li H, Mallick S, Dannemann M, Fu Q, Kircher M, Kuhlwilm M, Lachmann M, Meyer M, Ongyerth M, Siebauer M, Theunert C, Tandon A, Moorjani P, Pickrell J, Mullikin JC, Vohr SH, Green RE, Hellmann I, Johnson PL, Blanche H, Cann H, Kitzman JO, Shendure J, Eichler EE, Lein ES, Bakken TE, Golovanova LV, Doronichev VB, Shunkov MV, Derevianko AP, Viola B, Slatkin M, Reich D, Kelso J, Pääbo S (18 December 2013). "The complete genome sequence of a Neanderthal from the Altai Mountains". Nature. 505 (7481): 43–49. Bibcode:2014Natur.505...43P. doi:10.1038/nature12886. PMC . PMID 24352235.
- Callaway, Ewen (4 December 2013). "Hominin DNA baffles experts". Nature. 504 (7478): 16–17. Bibcode:2013Natur.504...16C. doi:. PMID 24305130.
- Zimmer, Carl (16 November 2015). "In a Tooth, DNA From Some Very Old Cousins, the Denisovans". نيويورك تايمز. مؤرشف من الأصل في 29 أبريل 202016 نوفمبر 2015.
- Sawyer, Susanna; Renaud, Gabriel; Viola, Bence; Hublin, Jean-Jacques; Gansauge, Marie-Theres; Shunkov, Michael V.; Derevianko, Anatoly P.; Prüfer, Kay; Kelso, Janet (11 November 2015). "Nuclear and mitochondrial DNA sequences from two Denisovan individuals". PNAS. 112 (51): 15696–700. Bibcode:2015PNAS..11215696S. doi:10.1073/pnas.1519905112. PMID 26630009.
- Higuchi, Russell; Bowman, Barbara; Freiberger, Mary; Ryder, Oliver A.; Wilson, Allan C. (15 November 1984). "DNA sequences from the quagga, an extinct member of the horse family". Nature. 312 (5991): 282–284. doi:10.1038/312282a0. PMID 6504142.
- "Genetic history of an archaic hominin group from Denisova Cave in Siberia". Nature. 468 (7327): 1053–1060. 23 December 2010. doi:10.1038/nature09710. PMID 21179161.
- Pennisi, E. (16 May 2013). "More Genomes From Denisova Cave Show Mixing of Early Human Groups". Science. 340 (6134): 799. doi:10.1126/science.340.6134.799. PMID 23687020.
- Mounier, Aurélien; Marchal, François; Condemi, Silvana (March 2009). "Is Homo heidelbergensis a distinct species? New insight on the Mauer mandible". Journal of Human Evolution. 56 (3): 219–246. doi:10.1016/j.jhevol.2008.12.006. PMID 19249816.
- Cartmill, Matt & Smith, Fred H. (2009). The Human Lineage. جون وايلي وأولاده . . مؤرشف من الأصل في 17 مايو 202021 أبريل 2013.
- Meyer, Matthias; Fu, Qiaomei; Aximu-Petri, Ayinuer; Glocke, Isabelle; Nickel, Birgit; Arsuaga, Juan-Luis; Martínez, Ignacio; Gracia, Ana; de Castro, José María Bermúdez (4 December 2013). "A mitochondrial genome sequence of a hominin from Sima de los Huesos" ( كتاب إلكتروني PDF ). Nature. 505 (7483): 403–406. doi:10.1038/nature12788. PMID 24305051. مؤرشف من الأصل ( كتاب إلكتروني PDF ) في 12 فبراير 201423 أبريل 2014.
- Zimmer, Carl. "Bringing Extinct Species Back To Life". National Geographic. مؤرشف من الأصل في 12 ديسمبر 201626 مارس 2014.
- Gray, Richard (31 Jan 2009). "Extinct ibex is resurrected by cloning". Telegraph. مؤرشف من الأصل في 13 نوفمبر 201409 أبريل 2014.
- Church, George. "Multiplex Automated Genomic Engineering (MAGE): A machine that speeds up evolution is revolutionizing genome design". Wyss Institute. مؤرشف من الأصل في 12 أكتوبر 201626 مارس 2014.
- The Editors (14 May 2013). "Why Efforts to Bring Extinct Species Back from the Dead Miss the Point". Scientific American. مؤرشف من الأصل في 17 مايو 202009 أبريل 2014.
- Pimm, S. L.; Russell, G. J.; Gittleman, J. L.; Brooks, T. M. (21 July 1995). et al 1995_Future of Biodiversity.pdf "The Future of Biodiversity" ( كتاب إلكتروني PDF ). Science. 269 (5222): 347–350. doi:10.1126/science.269.5222.347. PMID 17841251. مؤرشف من الأصل ( كتاب إلكتروني PDF ) في 13 أبريل 201409 أبريل 2014.
- Hogan, Michael. "Causes of Extinction". The Encyclopedia of Earth. مؤرشف من الأصل في 21 أبريل 201609 أبريل 2014.
- Church, George (20 August 2013). "De-Extinction Is a Good Idea". Scientific American. مؤرشف من الأصل في 30 أغسطس 201809 أبريل 2014.
- "Method: Principles". Amino Acid Geochronology Laboratory. Northern Arizona University. مؤرشف من الأصل في 14 مارس 201226 مارس 2014.
- Poinar, H. N.; Hoss, M.; Bada, J. L.; Paabo, S. (10 May 1996). "Amino Acid Racemization and the Preservation of Ancient DNA". Science. 272 (5263): 864–866. doi:10.1126/science.272.5263.864. PMID 8629020.
- Henn, B. M.; Gignoux, C. R.; Feldman, M. W.; Mountain, J. L. (6 October 2008). "Characterizing the Time Dependency of Human Mitochondrial DNA Mutation Rate Estimates". Molecular Biology and Evolution. 26 (1): 217–230. doi:10.1093/molbev/msn244. PMID 18984905.
- "Mitochondrial DNA". DNA Analyst Training. NFSTC Science Serving Justice. مؤرشف من الأصل في 01 فبراير 201423 أبريل 2014.
- Lang, BF; Gray, MW; Burger, G (1999). "Mitochondrial genome evolution and the origin of eukaryotes". Annual Review of Genetics. 33: 351–97. doi:10.1146/annurev.genet.33.1.351. PMID 10690412.
- Waggoner, Ben. "Molecular Palaeontology" ( كتاب إلكتروني PDF ). Nature Publishing Group. مؤرشف من الأصل ( كتاب إلكتروني PDF ) في 05 مارس 2016.
- Tagliavia, Marcello; Massa, Bruno; Albanese, Ida; La Farina, Mario (29 March 2011). "DNA Extraction From Orthoptera Museum Specimens" ( كتاب إلكتروني PDF ). Analytical Letters. 44 (6): 1058–1062. doi:10.1080/00032719.2010.506939. مؤرشف من الأصل ( كتاب إلكتروني PDF ) في 18 مايو 2020.
- Cano, RJ; Poinar, HN (Sep 1993). "Rapid isolation of DNA from fossil and museum specimens suitable for PCR". BioTechniques. 15 (3): 432–4, 436. PMID 8217155.
- S. Blair Hodges; Mary H. Schweitzer. Molecular Paleontology ( كتاب إلكتروني PDF ). Encyclopedia of Paleontology. صفحات 752–754. مؤرشف من الأصل ( كتاب إلكتروني PDF ) في 08 نوفمبر 2016.
- Tuross, N (1994). "The biochemistry of ancient DNA in bone". Experientia. 50 (6): 50:530–535. doi:10.1007/bf01921721. PMID 7517371.
- Tuross, N; Stathoplos, L (1993). Ancient proteins in fossil bones. Methods in Enzymology. 224. صفحات 121–129. doi:10.1016/0076-6879(93)24010-r. . PMID 8264383.