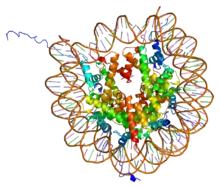
Les histones sont des protéines localisées dans le noyau des cellules eucaryotes[1] et dans les archées. Elles sont les principaux constituants protéiques des chromosomes. Elles sont en effet étroitement associées à l’ADN dont elles permettent la compaction, cette action formant des structures appelées nucléosomes : l'ADN est enroulé autour des histones comme du fil autour d'une bobine. Les histones sont très riches en acides aminés basiques (lysine et arginine), dont la charge positive à pH physiologique permet une interaction forte avec les groupements phosphate de l'ADN qui portent des charges négatives.
Structure
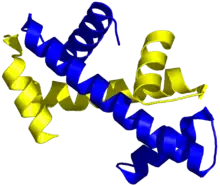
Les histones sont de petites protéines basiques de masse moléculaire comprise entre 13 et 15 kDa. Elles sont caractérisées par un domaine C-terminal globulaire, le domaine histone-fold. Ce domaine, très conservé depuis les archées jusqu’aux eucaryotes supérieurs, consiste en trois hélices α séparées par deux courtes boucles[2]. Il permet la dimérisation des histones selon un motif dit en poignée de main, qui sert de base à l’assemblage du nucléosome.
Le domaine histone-fold est retrouvé dans de nombreuses protéines autres que les histones, et définit la famille des protéines dite histone-like[3].
Les extrémités N-terminales des histones ne sont pas structurées et dépassent à l'extérieur de l'ADN dans le nucléosome assemblé. Ces extrémités sont accessibles à des enzymes de modification.
Différents types d'histones
Les histones comprennent cinq classes de protéines, regroupées en deux catégories en fonction de leur rôle dans la formation du nucléosome :
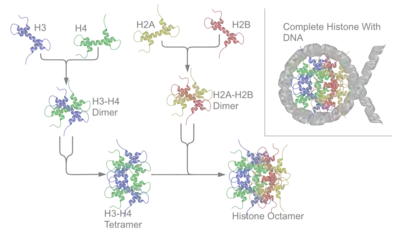
- les histones des classes H2A, H2B, H3 et H4 constituent les histones « de cœur », ainsi nommées car elles forment la particule de cœur du nucléosome : deux histones de chaque classe s’associent en un octamère autour duquel s’enroule le double-brin d’ADN sur environ cent cinquante paires de bases ;
- les histones de la classe H1 sont les histones dites « de liaison » : une histone de cette classe lie l’ADN à l’endroit où celui-ci entre et sort de la particule de cœur, et « scelle » ainsi le nucléosome[4].
Chaque classe comprend plusieurs sous-types (excepté la classe H4) distingués en fonction de leur profil d’expression :
- les sous-types dont l’expression est couplée à la réplication de l’ADN ;
- les sous-types dont l’expression est indépendante de la réplication de l’ADN ;
- les sous-types exprimés spécifiquement dans certains tissus ou à certaines étapes du développement.
Dans les cellules cyclantes, les sous-types dont l’expression est couplée à la réplication sont majoritaires dans la chromatine, c’est pourquoi on les qualifie d’histones conventionnelles ou canoniques. Par opposition, les autres sous-types sont qualifiés de ''variants d’histones'' ; ils représentent généralement moins de 10 % des histones totales dans les cellules cyclantes, mais cette proportion peut atteindre 50 % dans les cellules différenciées[5],[6].
Les gènes codant les histones conventionnelles sont généralement présents dans le génome en de multiples copies organisées en clusters — par exemple chez l’homme, trois clusters sur les chromosomes 1 et 6 ; chez la souris, trois clusters sur les chromosomes 3, 11 et 13[7]. Ils présentent des caractéristiques atypiques pour des gènes eucaryotes, comme l’absence d’introns et une terminaison de la transcription signalée par une structure de type tige-boucle au lieu d’un signal de polyadénylation. À l’opposé, les gènes codants les variants d’histones ne sont présents qu’en une ou deux copies et sont répartis isolément dans tout le génome[8] ; Ils possèdent tous au moins un intron et un signal de poly-adénylation.
Histone et condensation de l’ADN

Il y a environ cinquante-quatre paires de bases entre deux nucléosomes, cette valeur variant selon les espèces (par exemple, on en compte cent soixante-cinq pour la levure). Le niveau suivant de compaction de l'ADN fait intervenir d'autres protéines dites « non histones. »
Le degré de condensation de l'ADN autour des nucléosomes d'histones et des protéines non histones est variable le long des chromosomes, dans la chromatine. Il est faible dans l'euchromatine que l'on dit « ouverte » et accessible à la machinerie des ARN polymérases. Il est élevé dans l'hétérochromatine, que l'on dit « fermée » et « inaccessible » à la machinerie de transcription.
Ce degré de condensation est regulé par des modifications des extrémités N-terminales des histones, comme des phosphorylations, acétylations, méthylations, ubiquitinations, sumoylations, etc. l'ensemble de ces modifications étant catalysées par des enzymes spécifiques. Les modifications covalentes des histones agiraient soit directement en modifiant la compaction de l'enroulement d'ADN autour des nucléosomes, soit indirectement en constituant des « marques » permettant le recrutement de protéines capables de modifier la structure de la chromatine. Le modèle des modifications covalentes des histones agissant comme un code (le « code des histones ») a été proposé par Strahl et Allis en 2000 dans la revue Nature[9]. Cependant, ce code est, semble-t-il, loin d'être universel et plutôt relativement spécifique selon les gènes et les cellules considérés.
Code des histones
Le code des histones établit un lien direct entre la modification de certains résidus de la queue des histones qui crée des liaisons pour des effecteurs protéiques et l’état transcriptionnel de la chromatine[1].
Une modification d’histone peut en influencer une autre de manière synergique ou antagoniste ; c’est un mécanisme qui génère et stabilise des empreintes spécifiques.
L’acétylation (ajout de groupement acétyl) s’effectue sur certains résidus lysine précis par des enzymes nommées histones acétyl transférases (HAT).
Elle diminue généralement l’interaction inter-nucléosomes et entre les queues des histones et le fragment d’ADN qui fait le lien entre les nucléosomes. Ceci entraîne le relâchement de la chromatine, la faisant passer à l'état euchromatinien, et permet ainsi une meilleure accessibilité aux autres facteurs. L’acétylation est associée à une activation de la transcription et est facilement réversible grâce à l’action des histones désacétylases (HDAC).
La méthylation, quant à elle, peut s’effectuer soit sur des lysines soit sur des arginines et peut consister en l'ajout d'un, de deux ou de trois groupements méthyls.
Selon les résidus méthylés et le nombre de groupement ajouté elle est associée à une activation ou une répression de la transcription. Longtemps considérée comme statique, la méthylation des histones s'avère être une modification réversible impliquée dans un processus dynamique, bien que plus stable que l'acétylation et la phosphorylation. Un nombre croissant d'histones déméthylases sont identifiés[10].
De manière générale, ces deux types de modifications sont antagonistes, et la désacétylation des lysines doit précéder leur méthylation. Cet antagonisme entraine la mise en place d'un certain équilibre dynamique entre les territoires hétérochromatiniens (généralement non exprimables et méthylés sur certains acides aminés clés) et euchromatiniens (généralement exprimables et acétylés). Par exemple, la Lysine 9 de l'histone H3 est connue pour être associée à une répression de la chromatine environnante lorsqu'elle est méthylée. Cette méthylation est reconnue par une protéine, HP1, qui se fixe donc sur H3 méthylée. À son tour, HP1 attire la protéine Suv39, une Histone MethylTransférase, qui pourra méthyler la lysine 9 de l'histone H3 du nucléosome voisin, et ainsi de suite. On voit donc, comment, de proche en proche, les histones H3 seront méthylées et la chromatine sera condensée. Cependant, cette invasion hétérochromatinienne sera stoppée si la lysine 9 de H3 rencontrée est déjà acétylée. Ainsi se met en place un équilibre compétitif entre domaines chromatiniens exprimés et réprimés.
Les modifications des queues d'histones jouent le rôle de « marques » épigénétiques qui entraînent le recrutement de différentes classes de protéines, puisque les lysines acétylées ou méthylées sont reconnues par des domaines protéiques différents. De plus, le recrutement de certains facteurs au niveau de la chromatine nécessite l’existence préalable de modifications d’histones et de protéines déjà liées. Le code des histones est donc interprété dans le contexte d’autres facteurs associés à la chromatine et c’est la combinaison d’interaction entre les histones modifiées et d’autres facteurs qui détermine si une protéine est recrutée à la chromatine.
Variants d'histones
Dans plusieurs espèces eucaryotes, des variants d’histones, aussi appelés histones non canoniques, ont été découverts.
Ces variants ont une séquence qui diffère de celle des histones conventionnelles sur quelques résidus seulement (cas des variants dits homéomorphes), ou sur des portions plus importantes de la protéine (cas des variants hétéromorphes).
Les variants d’histones jouent des rôles majeurs dans différents aspects de la biologie tels que la réparation de l’ADN[11],[12], l’organisation centromérique[13], l’inactivation du chromosome sexuel X[14] et une condensation spécifique des cellules gamètes mâles[15],[16].
Histones et température
Chez l’arabette (Arabidopsis thaliana) une seule histone (H2A.Z) suffit à rendre ce taxon sensible des variations de température de moins de 1 °C. Cette histone modifie l’enroulement de l’ADN sur lui-même et contrôle ainsi l’accès à l’ADN de certaines molécules inhibant ou activant plusieurs dizaines de gènes. Cet effet « bio-thermostat » semble fréquent dans la nature, car également détecté chez la levure[17],[18].
Dans les spermatozoïdes
Les histones sont remplacées par des protamines, protéines riches en arginine et en cystéine.
La richesse en cystéine permet la formation de pont disulfure. Cette structure protège l'ADN lors d'éventuels déplacements liés à la fécondation.
Notes et références
- 1 2 Annabelle Gérard, Sophie Polo et Geneviève Almouzni, « Nom de code : histones », Pour la science, no 46, (ISSN 0153-4092, lire en ligne)
- ↑ Arents et Moudrianakis 1995
- ↑ Sullivan et al. 2002
- ↑ Fan et Roberts 2006
- ↑ Brush et al. 1985
- ↑ Wells et Kedes 1985
- ↑ Marzluff et al. 2002
- ↑ Kamakaka et Biggins 2005
- ↑ Strahl et Allis 2000
- ↑ Klose et Zhang 2007
- ↑ Redon et al. 2002
- ↑ Billon et Cote 2011
- ↑ Foltz et al. 2009
- ↑ Fernandez-Capetillo et al. 2003
- ↑ Okada et al. 2005
- ↑ Govin et al. 2004
- ↑ Kumar et Wigge 2010
- ↑ Perrier 2010
Voir aussi
Bibliographie
- (en) Gina Arents et Evangelos N. Moudrianakis, « The histone fold: a ubiquitous architectural motif utilized in DNA compaction and protein dimerization », Proc. Natl. Acad. Sci. USA, vol. 92, no 24, , p. 11170–11174 (PMID 7479959, lire en ligne [PDF])
- (en) David Brush, Jerry B. Dodgson, Ok-Ryun Choi, Priscilla Stevens et James Douglas Engel, « Replacement variant histone genes contain intervening sequences », Mol. Cell. Biol., vol. 5, no 6, , p. 1307–1317 (PMID 2863747, lire en ligne)
- (en) Li Fan et Victoria Roberts, « Complex of linker histone H5 with the nucleosome and its implications for chromatin packing », Proc. Natl. Acad. Sci. USA, vol. 103, no 22, , p. 8384–8389 (PMID 16717183, DOI 10.1073/pnas.0508951103, lire en ligne)
- (en) Oscar Fernandez-Capetillo, Shantha K. Mahadevaiah, Arkady Celeste, Peter J. Romanienko, R. Daniel Camerini-Otero, William M. Bonner, Katia Manova, Paul Burgoyne et André Nussenzweig, « H2AX is required for chromatin remodeling and inactivation of sex chromosomes in male mouse meiosis », Dev. Cell, vol. 4, no 4, , p. 497–508 (PMID 12689589)
- (en) Daniel Foltz, Lars Jansen, Aaron Bailey, John Yates, Emily Bassett, Stacey Wood, Ben Black et Don Cleveland, « Centromere-specific assembly of CENP-A nucleosomes is mediated by HJURP », Cell, vol. 137, no 3, , p. 472–484 (PMID 19410544, DOI 10.1016/j.cell.2009.02.039, lire en ligne)
- (en) Jérôme Govin, Cécile Caron, Cécile Lestrat, Sophie Rousseaux et Saadi Khochbin, « The role of histones in chromatin remodelling during mammalian spermiogenesis », Eur. J. Biochem., vol. 271, no 17, , p. 3459–3469 (PMID 15317581, DOI 10.1111/j.1432-1033.2004.04266.x, lire en ligne)
- (en) Rohinton Kamakaka et Sue Biggins, « Histone variants: deviants? », Genes Dev., vol. 19, no 3, , p. 295–310 (PMID 15687254, DOI 10.1101/gad.1272805, lire en ligne)
- (en) Robert Klose et Yi Zhang, « Regulation of histone methylation by demethylimination and demethylation », Nat. Rev. Mol. Cell Biol., vol. 8, no 4, , p. 307–318 (PMID 17342184, DOI 10.1038/nrm2143)
- (en) S. Vinod Kumar et Philip A. Wigge, « H2A.Z-containing nucleosomes mediate the thermosensory response in Arabidopsis », Cell, vol. 140, no 1, , p. 136–147 (PMID 20079334, DOI 10.1016/j.cell.2009.11.006)
- (en) William Marzluff, Preetam Gongidi, Keith Woods, Jianping Jin et Lois Maltais, « The human and mouse replication-dependent histone genes », Genomics, vol. 80, no 5, , p. 487–498 (PMID 12408966)
- (en) Takashi Okada, Makoto Endo, Mohan Singh et Prem Bhalla, « Analysis of the histone H3 gene family in Arabidopsis and identification of the male-gamete-specific variant AtMGH3 », Plant J., vol. 44, no 4, , p. 557–568 (PMID 16262706, DOI 10.1111/j.1365-313X.2005.02554.x)
- Jean-Jacques Perrier, « Une protéine thermomètre chez les plantes », Pour la science, (lire en ligne)
- (en) Christophe Redon, Duane Pilch, Emmy Rogakou, Olga Sedelnikova, Kenneth Newrock et William Bonner, « Histone H2A variants H2AX and H2AZ », Curr. Opin. Genet. Dev., vol. 12, no 2, , p. 162–169 (PMID 11893489)
- (en) Pierre Billon et Jacques Côté, « Precise deposition of histone H2A.Z in chromatin for genome expression and maintenance », Biochim Biophys Acta., (PMID 22027408, lire en ligne)
- (en) Brian D. Strahl et C. David Allis, « The language of covalent histone modifications », Nature, vol. 403, no 6765, , p. 41–45 (PMID 10638745, DOI 10.1038/47412)
- (en) Steven Sullivan, Daniel Sink, Kenneth Trout, Izabela Makalowska, Patrick Taylor, Andreas Baxevanis et David Landsman, « The Histone Database », Nucleic Acids Res., vol. 30, no 1, , p. 341–342 (PMID 11752331, lire en ligne)
- (en) Dan Wells et Larry Kedes, « Structure of a human histone cDNA: evidence that basally expressed histone genes have intervening sequences and encode polyadenylylated mRNAs », Proc. Natl. Acad. Sci. U.S.A., vol. 82, no 9, , p. 2834–2838 (PMID 2859593, lire en ligne [PDF])