إن فيروسات الأورام الحليمية هي عائلة تصنيفية قديمة من فيروسات الدنا غير المغلفة والمعروفة مجتمعة باسم فيروسات الورم الحليمي. [1] تم تحديد عدة مئات من أنواع فيروسات الورم الحليمي والتي يشار إليها تقليديًا باسم "الأنواع" [2] والتي تصيب جميع الثدييات التي تم فحصها بعناية وتصيب الفقاريات الأخرى مثل الطيور والثعابين والسلاحف والأسماك. [3] [4] [5] تعتمد العدوى من قبل معظم أنواع فيروس الورم الحليمي اعتمادًا على النوع فتكون إما بدون أعراض (مثل مبيتا- PVs وعظم بيتا) أو تسبب أورامًا حميدة صغيرة تعرف باسم الورم الحليمي أو الثآليل (مثل فيروس الورم الحليمي البشري 1 ، HPV6 أو HPV11). ومع ذلك فإن الأورام الحليمية التي تسببها بعض الأنواع ، مثل فيروس الورم الحليمي البشري 16 و 18 ، تحمل خطر الإصابة بالسرطان . [6]
ويعتبر الورم الحلمي عادة غاية المضيف- ونسيجيا مدار ويعتقد أنها نادرا ما تنتقل بين الأنواع. [7] وتتكاثر فيروسات الأورام الحليمية تكرار حصرا في الطبقة القاعدية من أنسجة سطح الجسم . وتصيب جميع أنواع فيروسات الورم الحليمي المعروفة سطحًا معينًا من الجسم [2] وعادةً الجلد أو الظهارة المخاطية للأعضاء التناسلية أو الشرج أو الفم أو الشعب الهوائية. [8] على سبيل المثال فيروس الورم الحليمي البشري (HPV) نوع 1 يميل لتصيب باطن القدمين وفيروس الورم الحليمي البشري من النوع 2 في راحتي اليدين حيث أنها قد تسبب الثآليل بالإضافة إلى ذلك هناك أوصاف لوجود الحمض النووي لفيروس الورم الحليمي في الدم وفي الخلايا أحادية النواة في الدم المحيطي .
حُدّدَت فيروسات الورم الحليمي لأول مرة في أوائل القرن العشرين عندما تبين أن الثآليل الجلدية أو الأورام الحليمية يمكن أن تنتقل بين الأفراد عن طريق عامل معدي قابل للتصفية. وفي عام 1935 تابع فرانسيس بيتون روس الذي أظهر سابقًا وجود فيروس ساركوما مسبب للسرطان في الدجاج ليثبت أن فيروس الورم الحليمي يمكن أن يسبب سرطان الجلد في الأرانب المصابة وكان هذا أول دليل على أن الفيروس يمكن أن يسبب سرطان الثدييات.
تصنيف فيروسات الورم الحليمي
إن هناك أكثر من 100 نوع من فيروس الورم الحليمي المعترف به [9] وبالرغم من أن الـ ITV تعترف رسميًا بعدد أصغر مصنف في 53 جنسًا اعتبارًا من عام 2019. [10] [11] [12] وجميع فيروسات الورم الحليمي (PVS) لها منظمات جينومية متشابهة وأي زوج من PVs يحتوي على خمسة جينات متماثلة على الأقل على الرغم من أن تسلسل النوكليوتيدات قد يتباعد بأكثر من 50 ٪. أدت خوارزميات التطور الجيني التي تسمح بمقارنة التماثلات إلى أشجار تطور جيني لها طوبولوجيا مماثلة ، مستقلة عن الجين الذي تم تحليله.
وتشير دراسات التطور الوراثي بقوة إلى أن الخلايا الكهروضوئية تتطور عادةً جنبًا إلى جنب مع الأنواع المستضيفة من الثدييات والطيور ولا تغير الأنواع المضيفة ولا تتجمع وقد حافظت على تنظيمها الجينومي الأساسي لفترة تتجاوز 100 مليون سنة. وأرست هذه المقارنات التسلسلية الأساس لتصنيف PV والذي أصبح الآن معترفاً به رسمياً من قبل اللجنة الدولية لتصنيف الفيروسات . إن جميع PVs تشكل عائلة فيروسات الأورام الحليمية، والتي تختلف عن فيروسات الأورام الحليمية وبالتالي القضاء على مصطلح فيروسات الأورام الحليمية . وتعتبر الفروع الرئيسية لشجرة السلالات من PVs أجناسًا و يتم تحديدها بالحروف اليونانية. وتعتبر الفروع الصغيرة أنواعًا وتوحد الأنواع الكهروضوئية المميزة جينيًا دون إظهار اختلافات بيولوجية معروفة. ولا يؤثر هذا النظام التصنيفي الجديد على التحديد والتوصيف التقليديين "للأنواع" الكهروضوئية وعزلها المستقل مع اختلافات جينومية بسيطة و يشار إليها باسم "الأنواع الفرعية" و "المتغيرات" وكلها تصنيفات أقل من مستوى "الأنواع". [13]
وقد يحتاج هذا التصنيف إلى مراجعة في ضوء وجود المؤتدات المؤتلفة لفيروس الورم الحليمي - الورم الحليمي. [14] كما تم وصف أنواع إضافية. تم عزل فيروس Sparus aurata papillomavirus 1 من الأسماك. [15]
فيروس الورم الحليمي البشري
أُجري تسلسل كامل لأكثر من 170 نوعًا من أنواع فيروس الورم الحليمي البشري. [16] وقد تم تقسيمها إلى 5 أجناس: Alphapapillomavirus و Betapapillomavirus و Gammapapillomavirus و Mupapillomavirus و Nupapillomavirus. وتم تحديد 200 فيروس إضافي على الأقل تنتظر التسلسل والتصنيف.
فيروس الورم الحليمي الحيواني
تميل أنواع فيروس الورم الحليمي الفردي إلى التكيف بشكل كبير مع التكاثر في نوع حيواني واحد. قام الباحثون في إحدى الدراسات بمسح جلد الجبين لمجموعة متنوعة من حيوانات حديقة الحيوان واستخدموا PCR لتضخيم أي DNA فيروس الورم الحليمي قد يكون موجودًا. [17] وعلى الرغم من تحديد مجموعة متنوعة من متواليات فيروس الورم الحليمي في الدراسة الا أنه لم يجد الباحثون سوى القليل من الأدلة لانتقال العدوى بين الأنواع. وتم العثور على أحد حراس حديقة الحيوان ليكون إيجابيًا بشكل عابر لتسلسل فيروس الورم الحليمي الخاص بالشمبانزي. ومع ذلك لاحظ المؤلفون أن تسلسل فيروس الورم الحليمي الخاص بالشمبانزي يمكن أن يكون نتيجة تلوث سطح جلد حارس حديقة الحيوان ، على عكس العدوى المنتجة.
ويمكن أن يسبب فيروس الورم الحليمي للأرنب القطني (CRPV) ثآليل منتجة في مضيفه الأصلي في جنس الأرانب في أمريكا الشمالية Sylvilagus .و قد تكون هذه الثآليل التي تشبه القرن هي الأساس الأصلي للأساطير الحضرية للأرنب الأمريكي ذو القرون Jackalope و European Wolpertinger . [18] ويمكن أن تصاب الأرانب المحلية الأوروبية (جنس Oryctolagus ) بشكل عابر بفيروس CRPV في بيئة المختبر ، و نظرًا لأن الأرانب المحلية الأوروبية لا تنتج فيروسًا ذريًا معديًا فإنها تعتبر مضيفًا عرضيًا أو "مسدودًا" لـ CRPV. [19]
كما تم توثيق الانتقال بين الأنواع لفيروس الورم الحليمي البقري من النوع 1. [20] في مضيفه الطبيعي (الماشية) يحرض BPV-1 ثآليل جلدية ليفية كبيرة. ويمكن أن تؤدي عدوى BPV-1 للخيول وهي مضيف عرضي للفيروس و إلى تطور أورام حميدة تعرف باسم الساركويد .و حفزت الأهمية الزراعية لـ BPV-1 جهدًا ناجحًا لتطوير لقاح ضد الفيروس.
وقد حددت بعض التقارير فيروسات الورم الحليمي في القوارض الأصغر مثل الهامستر السوري والفأر الأفريقي المتعدد الثدييات والفأر الأوراسي . [21] ومع ذلك لا توجد فيروسات حلقية معروفة بأنها قادرة على إصابة فئران المختبر. وكان الافتقار إلى نموذج فأري قابل للتتبع لعدوى فيروس الورم الحليمي (بابيلوما) أحد القيود الرئيسية على الفحص المختبري لفيروسات الورم الحليمي.
ومن المعروف أن أربعة فيروسات حُليميَّة تُصيب الطيور: Fringilla coelebs papillomavirus 1، Francolinus leucoscepus papillomavirus 1، Psittacus erithacus papillomavirus 1 and Pygoscelis adeliae papillomavirus 1. [22] كل هذه الأنواع لها جينة (E9) ذات وظيفة غير معروفة مما يشير إلى أصل مشترك.
تطور
ويعتقد أن تطور فيروسات الورم الحليمي بطيء مقارنة بالعديد من أنواع الفيروسات الأخرى ولكن لا توجد قياسات تجريبية متاحة حاليًا. وربما يرجع ذلك إلى أن جينوم فيروس الورم الحليمي يتكون من حمض نووي مزدوج الجديلة مستقر وراثيًا والذي يتم نسخه بدقة عالية بواسطة آلة نسخ الحمض النووي للخلية المضيفة.
يعتقد أن فيروسات الورم الحليمي تتطور بشكل عام مع نوع معين من الحيوانات المضيفة على مدى سنوات عديدة على الرغم من وجود أدلة قوية ضد فرضية التطور المشترك. [23] وفي مثال سريع بشكل خاص ، تطورت HPV-16 بشكل طفيف مع توسع السكان في جميع أنحاء العالم وتختلف الآن في مناطق جغرافية مختلفة بطريقة تعكس على الأرجح تاريخ الهجرة البشرية. [24] [25]
تختلف أنواع فيروس الورم الحليمي البشري الأخرى مثل HPV-13 ، قليلًا نسبيًا في مجموعات بشرية مختلفة. وفي الواقع يشبه تسلسل فيروس الورم الحليمي البشري 13 بشكل كبير فيروس الورم الحليمي من البونوبو (المعروف أيضًا باسم الشمبانزي الأقزام). [26] وليس من الواضح ما إذا كان هذا التشابه ناتجًا عن الانتقال الأخير بين الأنواع أو لأن فيروس الورم الحليمي البشري -13 لم يتغير إلا قليلاً جدًا في السنوات الست أو نحو ذلك منذ اختلاف البشر والبونوبو. [25]
ويقدر أن أحدث سلف مشترك لهذه المجموعة من الفيروسات كان موجودًا 424 . [27]
إن هناك خمسة أجناس رئيسية تصيب البشر وهي (ألفا ، بيتا ، جاما ، مو ونو). تطور أحدث سلف مشترك لهذه الأجناس 49.7 - 58.5 . [28] قُدِّر أن أحدث سلف لجاما قد تطور بين 45.3 و 67.5 .
بناء
الورم الحلمي غير قابلة لليلفها وهذا يعني أن القشرة الخارجية أو قفيصة الفيروس لا تغطيها الدهون غشاء . إن بروتين فيروسي واحد يُعرف باسم L1 ضروري وكافٍ لتشكيل كبسولة 55-60 نانومتر تتكون من 72 كبسولة على شكل نجمة (انظر الشكل). مثل معظم الفيروسات غير المغلفة يكون الغطاء هو المنتظم هندسيًا ويقدم تناظرًا متساوي السطوح . و إن الجسيمات الشبيهة بالفيروسات المجمعة ذاتيًا والمكونة من L1 هي أساس مجموعة ناجحة من لقاحات فيروس الورم الحليمي البشري الوقائي المصممة لاستحضار الأجسام المضادة المحايدة للفيروسات التي تحمي من الإصابة بفيروس الورم الحليمي البشري الأولي. على هذا النحو ، papillomaviridæ مستقرة في البيئة .
جينوم فيروس الورم الحليمي هو جزيء DNA دائري مزدوج الجدائل ~ 8000 زوج أساسي في الطول. ويتم تعبئتها داخل قشرة L1 مع بروتينات الهيستون الخلوية والتي تعمل على لف وتكثيف الحمض النووي.
تحتوي كبسولة فيروس الورم الحليمي أيضًا على بروتين فيروسي يعرف باسم L2 وهو أقل وفرة. وعلى الرغم من عدم وضوح كيفية ترتيب L2 داخل الفيروس فمن المعروف أنه يؤدي العديد من الوظائف المهمة بما في ذلك تسهيل تعبئة الجينوم الفيروسي في الفيروسات الوليدة وكذلك الدخول المعدي للفيروس إلى الخلايا المضيفة الجديدة و يعتبر L2 مفيدًا كهدف محتمل لقاحات HPV الواقية على نطاق أوسع.
وعلى الرغم من أن العديد من الفيروسات تقدم 72 كبسولة ، فإن الورم الحليمي فريد تقريبًا من حيث أن جميع كبسولاته مصنوعة من تفاعلات الخماسي بين البروتينات. [29] وتظهر الفيروسات الأيكوزاهيدرالية الأخرى بدقة 12 كبسولة خماسية فقط أي كبسولات أخرى ستكون تفاعلات بروتين سداسي.و لذلك كان الورم الحليمي أول استثناء معروف لـ "نظرية شبه التكافؤ" والتي تنص بشكل أساسي على أن أغطية الفيروس تشبه غولدبرغ متعدد الوجوه . وفي الواقع تبين أن تخطيط البروتين في الورم الحليمي لا يتوافق مع أي متعدد الوجوه الكروية المعروفة و لذلك كان هيكلها سؤالًا مفتوحًا لأكثر من عقدين. تم الرد على السؤال في نهاية المطاف في عام 2003 من قبل Reidun Twarock ، باستخدام نموذج يستند إلى بلاط بنروز [30] مع بروتينين في ديمر يحل محل المعين وثلاثة بروتينات في أداة تشذيب تحل محل طائرة ورقية.
خصوصية الأنسجة
تتكاثر فيروسات الورم الحليمي بشكل حصري في الخلايا الكيراتينية و تشكل الخلايا الكيراتينية الطبقات الخارجية من الجلد بالإضافة إلى بعض الأسطح المخاطية مثل داخل الخد أو جدران المهبل، و تتكون هذه الأنسجة السطحية والمعروفة باسم الظهارة الحرشفية الطبقية من طبقات مكدسة من الخلايا المسطحة و تتشكل طبقات الخلايا من خلال عملية تعرف باسم التمايز الخلوي حيث تصبح الخلايا الكيراتينية تدريجياً متخصصة وتشكل في النهاية سطحًا صلبًا متشابكًا يمنع فقدان الرطوبة ويعمل كحاجز ضد مسببات الأمراض.و يعتقد أن الخلايا الجذعية الكيراتينية الأقل تمايزًا التي تم تجديدها على الطبقة السطحية هي الهدف الأولي للعدوى المنتجة لفيروس الورم الحليمي. وتعتمد الخطوات اللاحقة في دورة الحياة الفيروسية بشكل صارم على عملية تمايز الخلايا الكيراتينية. ونتيجة لذلك يمكن أن تتكاثر فيروسات الورم الحليمي فقط في أنسجة سطح الجسم.
دورة الحياة
دخول معدي
يمكن لفيروسات الورم الحليمي الوصول إلى الخلايا الجذعية للخلايا الكيراتينية من خلال الجروح الصغيرة المعروفة باسم الصدمات الدقيقة في الجلد أو السطح المخاطي. وتعزز التفاعلات بين L1 والسكريات الكبريتية على سطح الخلية التعلق الأولي بالفيروس. [31] [32] وثم يتمكن الفيروس من الدخول من سطح الخلية عن طريق التفاعل مع مستقبلات معينة على الأرجح عبر alpha-6 beta-4 Integrin ، [33] [34] ونقله إلى الحويصلات المغلقة بغشاء تسمى إندوسومات . [35] [36] يعطل بروتين الكبسولة L2 غشاء الإندوسوم من خلال الببتيد الموجب المخترق للخلايا مما يسمح للجينوم الفيروسي بالهروب والمرور إلى جانب L2 إلى نواة الخلية. [37] [38] [39]
استمرار الفيروسية
بعد الإصابة الناجح لالقرنية فإن الفيروس يعبر عن E1 و E2 البروتينات والتي هي للتكرار والحفاظ على الحمض النووي الفيروسي كما تعميما يصبوغ . وتعمل الجينات الفيروسية E6 و E7 على تعزيز نمو الخلايا عن طريق تعطيل بروتينات مثبط الورم p53 و pRb . ويمكن للخلايا الجذعية الكيراتينية في الطبقة القاعدية الظهارية الحفاظ على جينومات فيروس الورم الحليمي لعقود. [8]
إنتاج فيروس النسل
ويقتصر التعبير عن الجينات الفيروسية المتأخرة L1 و L2 على تمييز الخلايا الكيراتينية في الطبقات الخارجية من الجلد أو سطح الغشاء المخاطي.و يرتبط التعبير المتزايد عن L1 و L2 عادةً بزيادة كبيرة في عدد نسخ الجينوم الفيروسي ويُعتقد أن هذا التقييد للتعبير الجيني المتأخر الفيروسي يمثل شكلاً من أشكال التهرب المناعي نظرًا لأن الطبقات الخارجية للظهارة الحرشفية الطبقية تخضع لمراقبة محدودة نسبيًا بواسطة خلايا الجهاز المناعي.
ويتم تجميع فيروسات النسل المعدية الجديدة في نواة الخلية . طورت فيروسات الورم الحليمي آلية لإطلاق الفيروسات في البيئة. وتستخدم أنواع أخرى من فيروسات الحيوانات غير المغلفة عملية تحويضية نشطة لقتل الخلية المضيفة مما يسمح بإطلاق جزيئات فيروس النسل. وغالبًا ما ترتبط هذه العملية الغدة بالالتهاب والتي قد تؤدي إلى هجوم مناعي ضد الفيروس. وفيروسات الورم الحليمي تستغل التقشر كآلية إطلاق خفية غير التهابية.
الارتباط بالسرطان
على الرغم من أن بعض أنواع فيروس الورم الحليمي يمكن أن تسبب السرطان في الأنسجة الظهارية التي يعيشون فيها فإن السرطان ليس نتيجة نموذجية للعدوى. وعادة ما يحدث تطور السرطانات التي يسببها فيروس الورم الحليمي على مدى سنوات عديدة. وارتبط فيروس الورم الحليمي بتطور سرطان عنق الرحم وسرطان القضيب [40] وسرطانات الفم . [41] كما لوحظ وجود ارتباط مع سرطان الفرج وسرطان الكلى مع التمايز الحرفي في المرضى الذين يعانون من المثانة العصبية. [42] [43] ويوجد سرطان يسبب فيروسات الورم الحليمي الذي يشفر بروتينين صغيرين يسمى E6 و E7 يحاكيان السرطان الذي يسبب الأورام طريقة عملها هي أنها تحفز النمو غير الطبيعي للخلايا وتحجب دفاعاتها الطبيعية. كما أنها تعمل على العديد من بروتينات الإشارات التي تتحكم في الانتشار والاستماتة. [44]
دراسة مخبرية
حقيقة أن دورة حياة فيروس الورم الحليمي تتطلب بدقة تمايز الخلايا الكيراتينية قد شكلت حاجزًا كبيرًا أمام دراسة فيروسات الورم الحليمي في المختبر حيث أنها تحولت دون استخدام خطوط الخلايا التقليدية لتنمية الفيروسات. ونظرًا لأنه يمكن استخراج فيروسات BPV-1 المعدية من الثآليل الكبيرة التي يسببها الفيروس على الماشية فقد كان نوعًا من أنواع فيروس الورم الحليمي النموذجي لسنوات عديدة. كما تم استخدام CRPV وفيروس الورم الحليمي عن طريق الفم (ROPV) وفيروس الورم الحليمي الفموي (COPV) على نطاق واسع للدراسات المختبرية. وبمجرد اكتشاف الباحثين أن هذه الفيروسات تسبب السرطان عملوا معًا لإيجاد لقاح له. وحاليًا إن الطريقة الأكثر فعالية لتحقيق ذلك هي محاكاة فيروس يتكون من بروتين L1 ولكنه يفتقر إلى الحمض النووي. وبشكل أساسي يقوم نظام المناعة لدينا ببناء دفاعات ضد العدوى ولكن إذا كانت هذه العدوى لا تسبب المرض فيمكن استخدامها كقاح. يوضح إدخال PDB 6bt3 كيف تهاجم أسطح الأجسام المضادة سطح الفيروس لتعطيله. [45]
وقد تم نشر بعض أنواع فيروس الورم الحليمي البشري المنقولة جنسيًا باستخدام نظام "xenograft" للماوس حيث يتم زرع الخلايا البشرية المصابة بفيروس الورم الحليمي البشري في الفئران التي تعاني من نقص المناعة . في الآونة الأخيرة نجحت بعض المجموعات في عزل فيروس الورم الحليمي البشري - 16 المعدية من آفات عنق الرحم البشرية. ومع ذلك فإن عزل الفيروسات المعدية باستخدام هذه التقنية أمر شاق وعائد الفيروسات المعدية منخفض جدًا.
يمكن محاكاة تمايز الخلايا الكيراتينية في المختبر عن طريق تعريض الخلايا الكيراتينية المستزرعة لواجهة الهواء / السائل. كان تكييف أنظمة "ثقافة الطوافة" هذه مع دراسة فيروسات الورم الحليمي اختراقاً هاماً في الدراسة المختبرية لدورة الحياة الفيروسية. [46] ومع ذلك فإن أنظمة تربية الطوافة مرهقة نسبيًا ويمكن أن يكون غلة فيروس الورم الحليمي البشري المعدي منخفضًا. [47]
يوفر تطوير نظام قائم على الخميرة يسمح بتكرار فيروس الورم الحليمي البشري المستقر وسيلة مريحة وسريعة وغير مكلفة لدراسة العديد من جوانب دورة حياة فيروس الورم الحليمي البشري (أنجليتي 2002). على سبيل المثال يمكن إعادة إنشاء النسخ المعتمدة على E2 ، وتضخيم الجينوم والتغليف الفعال للحمض النووي HPV كامل الطول بسهولة في الخميرة (أنجليتي 2005).
و تم تطوير طرق عابرة عالية الإنتاجية في الآونة الأخيرة لإنتاج فيروسات زائفة من فيروس الورم الحليمي البشري تحمل جينات مراسل. وعلى الرغم من أن الفيروسات الكاذبة ليست مناسبة لدراسة جوانب معينة من دورة الحياة الفيروسية وتشير الدراسات الأولية إلى أن هيكلها ودخولها المعدية الأولية إلى الخلايا ربما تكون مشابهة في العديد من الطرق لفيروسات الورم الحليمي الأصلي.
ويرتبط فيروس الورم الحليمي البشري بجزيئات الهيبارين على سطح الخلايا التي يصيبها. وقد أظهرت الدراسات أن بلورة L1 كبسولات المعزولة تحتوي على سلاسل الهيبارين التي تتعرف عليها خطوط أخاديد اللايسين على سطح الفيروس، أيضا أولئك الذين لديهم أجسام مضادة يظهرون أنه يمكنهم منع هذا الاعتراف. [48]
التنظيم الجيني والتعبير الجيني
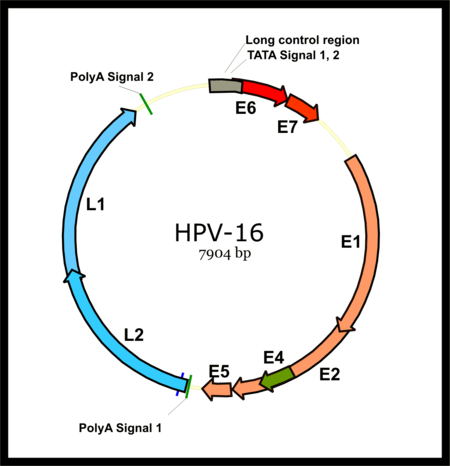
وينقسم جينوم فيروس الورم الحليمي إلى منطقة مبكرة (E) ، يشفر ستة إطارات قراءة مفتوحة (ORF) (E1 و E2 و E4 و E5 و E6 و E7) ويتم التعبير عنها فورًا بعد الإصابة الأولية لخلية مضيفة وآخر المنطقة (L) ترميز بروتين قابس رئيسي L1 وبروتين قابس صغير L2. ويتم ترميز جميع ORFs الفيروسية على حبلا DNA واحد (انظر الشكل). ويمثل هذا اختلافًا كبيرًا بين فيروسات الورم الحليمي والفيروسات المتعددة لأن نوع الفيروس الأخير يعبر عن جيناته المبكرة والمتأخرة عن طريق النسخ ثنائي الاتجاه لكل من خيوط الحمض النووي. وكان هذا الاختلاف عاملاً رئيسيًا في إنشاء الإجماع على أن فيروسات الورم الحليمي والفيروسات المتعددة ربما لم تشارك أبدًا سلفًا مشتركًا ، على الرغم من أوجه التشابه اللافتة في هياكل الفيروسات الخاصة بهم.
بعد إصابة الخلية المضيفة و يتم تنشيط المروج المبكر HPV16 ويتم نسخ الحمض النووي الريبي الأولي متعدد الكتل الذي يحتوي على جميع ORFs الستة المبكرة.و يحتوي هذا الحمض النووي الريبي متعدد الكتل على ثلاثة exons واثنان من الإنترونات ويخضع لربط RNA النشط لتوليد أشكال متعددة من mRNAs. [49] واحد من الحمض النووي الريبي الاسمي المقسم ، E6 * I ، بمثابة E7 mRNA لترجمة E7 oncoprotein. [50] وعلى النقيض من ذلك يلزم وجود إنترون في E6 ORF الذي لا يزال سليماً بدون ربط للترجمة من E6 oncoprotein. ومع ذلك فإن النسخ المبكر الفيروسي يخضع لتنظيم E2 الفيروسي ومستويات E2 العالية يقمع النسخ. وتتكامل جينومات فيروس الورم الحليمي البشري في الجينوم المضيف عن طريق تعطيل E2 ORF ، مما يمنع قمع E2 على E6 و E7. وبالتالي فإن تكامل الجينوم الفيروسي في جينوم DNA المضيف يزيد من تعبير E6 و E7 لتعزيز الانتشار الخلوي وفرصة الورم الخبيث.
ينشط المروج الفيروسي المتأخر الرئيسي في المنطقة المبكرة الفيروسية فقط في الخلايا المتمايزة ويمكن تعزيز نشاطه بشكل كبير عن طريق تضاعف الحمض النووي الفيروسي. إن النص المتأخر هو أيضا RNA متعدد الأوجه يحتوي على اثنين من الإنترونات وثلاثة exons. ويعتبر الربط RNA البديل لهذا النص المتأخر ضروريًا للتعبير L1 و L2 ويمكن تنظيمه بواسطة عناصر RIS وعناصر الربط المضيفة. [49] [51] [52]
المناقشة الفنية لوظائف الجين الورم الحليمي
وعادة ما يتم تحديد الجينات الموجودة في جينوم فيروس الورم الحليمي بعد التشابه مع الجينات الأخرى التي تم تحديدها مسبقًا. ومع ذلك قد تكون بعض إطارات القراءة المفتوحة الزائفة مخطئة على أنها جينات بعد وضعها في الجينوم وقد لا تكون جينات حقيقية. وينطبق هذا بشكل خاص على بعض إطارات القراءة المفتوحة E3 و E4 و E5 و E8.
E1
ويشفر بروتينًا يرتبط بالأصل الفيروسي للنسخ المتماثل في منطقة التحكم الطويلة للجينوم الفيروسي. ويستخدم E1 ATP لممارسة نشاط helicase الذي يفصل خيوط الحمض النووي وبالتالي تحضير الجينوم الفيروسي للنسخ بواسطة عوامل تضاعف DNA الخلوي.
E2
يعمل بروتين E2 كمنظم نسخ رئيسي للمروجين الفيروسيين الموجودين أساسًا في منطقة التحكم الطويلة. ,يحتوي البروتين على مجال معاملات مرتبط بمنطقة مفصلة غير منظمة نسبيًا بمجال ربط DNA مميز. يسهل E2 ربط E1 بالأصل الفيروسي للنسخ المتماثل. ويستخدم E2 أيضًا بروتينًا خلويًا يعرف باسم برومودومين -4 (Brd4) لربط الجينوم الفيروسي بالكروموسومات الخلوية. [53] ويضمن هذا الارتباط بالمصفوفة النووية للخلية التوزيع الأمين للجينوم الفيروسي لكل خلية ابنة بعد انقسام الخلية.و يُعتقد أن E2 يعمل كمنظم سلبي للتعبير عن الجينات الورمية E6 و E7 في الخلايا الكيراتينية المصابة بفيروس HPV. وتميل التغيرات الجينية مثل دمج الحمض النووي الفيروسي في كروموسوم الخلية المضيفة التي تعطل التعبير E2 إلى زيادة التعبير عن الجينات الورمية E6 و E7 مما يؤدي إلى التحول الخلوي وربما المزيد من زعزعة الاستقرار الجيني.
E3
هذا الجين المفترض الصغير موجود فقط في عدد قليل من أنواع فيروس الورم الحليمي. إن الجين لا يُعرف على أنه بروتين ولا يبدو أنه يؤدي أي وظيفة.
E4
وعلى الرغم من أن البروتينات E4 يتم التعبير عنها بمستويات منخفضة خلال المرحلة المبكرة من العدوى الفيروسية إلا أن التعبير عن E4 يزداد بشكل كبير خلال المرحلة المتأخرة من العدوى. وبعبارة أخرى ، فإن تسمية "E" قد تكون تسمية خاطئة. في حالة HPV-1 و يمكن أن يمثل E4 ما يصل إلى 30 ٪ من إجمالي البروتين على سطح الثؤلول. [54] ويعتقد أن بروتين E4 من العديد من أنواع فيروس الورم الحليمي لتسهيل الإفراج الفيريون في البيئة عن طريق تعطيل خيط متوسط من الخلايا الكيراتينية الهيكل الخلوي . إن الطفرات الفيروسية غير القادرة على التعبير عن E4 لا تدعم النسخ عالي المستوى للحمض النووي الفيروسي ولكن ليس من الواضح حتى الآن كيف يسهل E4 نسخ الحمض النووي. وكما ثبت أن E4 يشارك في القبض على الخلايا في مرحلة G2 من دورة الخلية .
E5
E5 هي بروتينات صغيرة مسعورة للغاية تعمل على زعزعة وظيفة العديد من بروتينات الغشاء في الخلية المصابة. [55] ويعمل بروتين E5 لبعض أنواع فيروس الورم الحليمي الحيواني (بشكل رئيسي نوع فيروس الورم الحليمي البقري 1) كورم سرطاني في المقام الأول عن طريق تنشيط إشارات تعزيز نمو الخلية لمستقبلات عامل النمو المشتق من الصفائح الدموية . ومع ذلك يبدو أن بروتينات E5 من فيروسات الورم الحليمي البشري المرتبطة بالسرطان تنشط سلسلة الإشارة التي بدأها عامل نمو البشرة عند ربط الليجاند. كما ثبت أن HPV16 E5 و HPV2 E5 يقللان من تنظيم التعبير السطحي لبروتينات الفئة الأولى المعقدة للتوافق النسيجي ، والتي قد تمنع الخلايا المصابة من التخلص من الخلايا التائية القاتلة .
E6

E6 هو 151 ببتيد من الأحماض الأمينية يشتمل على شكل من نوع 1 بتسلسل إجماعي - (T / S) - (X) - (V / I) -COOH. [57] [58] كما أن لديها زخارف إصبع من الزنك .
إن لـ E6 أهمية خاصة لأنه يبدو أن له أدوارًا متعددة في الخلية ويتفاعل مع العديد من البروتينات الأخرى. ومع ذلك فإن دورها الرئيسي هو التوسط في تدهور p53 وهو بروتين كبت ورم رئيسي مما يقلل من قدرة الخلية على الاستجابة لتلف الحمض النووي . [59] [60]
وقد ثبت أيضًا أن E6 يستهدف البروتينات الخلوية الأخرى وبالتالي يغير العديد من مسارات التمثيل الغذائي . إن أحد هذه الأهداف هو NFX1-91 ، الذي يقمع عادة إنتاج التيلوميراز وهو بروتين يسمح للخلايا بتقسيم عدد غير محدود من المرات. وعندما يتحلل NFX1-91 بواسطة E6 تزداد مستويات الإنزيم تيلوميراز مما يعطل آلية رئيسية تحافظ على نمو الخلايا تحت السيطرة. [61] وبالإضافة إلى ذلك يمكن أن يعمل E6 كعامل مساعد نسخي - على وجه التحديد ، منشط النسخ - عند التفاعل مع عامل النسخ الخلوي ، E2F1 / DP1. [57]
يمكن أيضًا ربط E6 بنطاقات PDZ وهي تسلسلات قصيرة غالبًا ما توجد في بروتينات الإشارة. ويسمح الزخم الهيكلي لـ E6 بالتفاعل مع نطاقات PDZ على جينات الورم DLG (الأقراص الكبيرة) و hDLG (ذبابة الفاكهة الكبيرة). [58] [62] ويؤدي الارتباط في هذه المواقع إلى تحويل بروتين DLG وتعطيل وظيفة الكابت. وتتفاعل بروتينات E6 أيضًا مع بروتينات MAGUK (عائلة كينيز الغانيلات المرتبطة بالغشاء) وهذه البروتينات بما في ذلك MAGI-1 و MAGI-2 و MAGI-3 هي عادة بروتينات هيكلية ويمكن أن تساعد في الإشارة. و الأهم من ذلك يعتقد أنهم متورطون في نشاط قمع DLG. وعندما تتجمع E6 مع نطاقات PDZ على بروتينات MAGI ، فإنها تشوه شكلها وبالتالي تعوق وظيفتها و بشكل عام يعمل بروتين E6 على إعاقة نشاط البروتين الطبيعي بطريقة تسمح للخلية بالنمو والتكاثر بمعدل زيادة السرطانات.
ونظرًا لأن التعبير عن E6 مطلوب بشكل صارم للحفاظ على النمط الظاهري الخبيث في السرطانات التي يسببها فيروس الورم الحليمي البشري فهو هدف جذاب لقاحات HPV العلاجية المصممة للقضاء على أورام سرطان عنق الرحم المعروفة.
E7
وفي معظم أنواع فيروسات الورم الحليمي تكون الوظيفة الأساسية لبروتين E7 هي تعطيل أعضاء عائلة pRb من بروتينات كابتة الورم و جنبا إلى جنب مع E6 و E7 يعمل على منع موت الخلايا ( موت الخلايا المبرمج ) وتعزيز تقدم دورة الخلية وبالتالي تحضير الخلية لتكرار الحمض النووي الفيروسي.و يشارك E7 أيضًا في تخليد الخلايا المصابة عن طريق تنشيط التيلوميراز الخلوي. مثل E6 و E7 هو موضوع اهتمام بحثي مكثف ويعتقد أنه يمارس مجموعة واسعة من التأثيرات الأخرى على الخلايا المصابة. وكما هو الحال مع E6 فإن التعبير المستمر عن E7 مطلوب لبقاء خطوط الخلايا السرطانية مثل HeLa المشتقة من الأورام التي يسببها فيروس الورم الحليمي البشري. [63]
E8
إن عدد قليل فقط من أنواع فيروسات الورم الحليمي ترميز بروتين قصير من الجين E8 وفي حالة BPV-4 (جنس فيروس الورم الحليمي Xi ) قد يحل إطار القراءة المفتوح E8 محل إطار القراءة المفتوح E6 وهو غير موجود في جنس فيروس الورم الحليمي هذا [64] وتشبه جينات E8 كيميائيًا ووظيفيًا جينات E5 من بعض فيروسات الورم الحليمي البشري وتسمى أيضًا E5 / E8.
L1
ويتجمع L1 تلقائيًا في كبسولات خماسية و يمكن أن تستمر الكبسولات النقية لتشكيل الكبسولات والتي يتم تثبيتها بواسطة روابط ثنائي الكبريتيد بين جزيئات L1 المجاورة. وتعتبر كبسولات L1 المجمعة في المختبر أساس اللقاحات الوقائية ضد العديد من أنواع فيروس الورم الحليمي البشري وبالمقارنة مع جينات فيروس الورم الحليمي الأخرى فإن تسلسلات الأحماض الأمينية لمعظم أجزاء L1 محفوظة بشكل جيد بين الأنواع ومع ذلك يمكن أن تختلف الحلقات السطحية لـ L1 بشكل كبير حتى بالنسبة لأعضاء مختلفين من أنواع معينة من فيروس الورم الحليمي.و من المحتمل أن يعكس هذا آلية للتهرب من استجابات الأجسام المضادة المحايدة الناتجة عن عدوى فيروس الورم الحليمي السابقة. [65]
L2
إن L2 موجود في حالة مؤكسدة داخل فيريوس الورم الحليمي حيث يشكل بقايا السيستين المحفوظين رابطة ثنائي كبريتيد داخل الجزيئات [66] بالإضافة إلى التعاون مع L1 لتعبئة الحمض النووي الفيروسي في الفيريون فقد ثبت أن L2 يتفاعل مع عدد من البروتينات الخلوية أثناء عملية الدخول المعدية بعد الربط الأولي للفيريون بالخلية و يجب تشريح L2 بواسطة فوران البروتياز الخلوي. [67] ويتم إدخال الفيريون على الأرجح من خلال عملية بوساطة الكاثرين إلى إندوسوم حيث يعتقد أن الظروف الحمضية تؤدي إلى التعرض لأجزاء من زعزعة الاستقرار في L2. [37] وقد تشارك أيضًا البروتينات الخلوية بيتا أكتين [68] و syntaxin -18 [69] في أحداث الدخول بوساطة L2. وبعد هروب الإندوسوم يتم استيراد L2 والجينوم الفيروسي إلى نواة الخلية حيث ينتقلان إلى مجال شبه نووي يعرف باسم جسم ND-10 الغني بعوامل النسخ . [38] ويتم حفظ أجزاء صغيرة من L2 بشكل جيد بين أنواع فيروس الورم الحليمي المختلفة وقد توفر اللقاحات التجريبية التي تستهدف هذه المجالات المحفوظة الحماية ضد مجموعة واسعة من أنواع فيروس الورم الحليمي البشري. [70]
مراجع
- Van Doorslaer, K; Chen, Z; Bernard, HU; Chan, PKS; DeSalle, R; Dillner, J; Forslund, O; Haga, T; McBride, AA (August 2018). "ICTV Virus Taxonomy Profile: Papillomaviridae". The Journal of General Virology. 99 (8): 989–990. doi:10.1099/jgv.0.001105. PMID 29927370.
- "Classification of papillomaviruses". Virology. 324 (1): 17–27. June 2004. doi:10.1016/j.virol.2004.03.033. PMID 15183049.
- "Genomic characterization of two novel reptilian papillomaviruses, Chelonia mydas papillomavirus 1 and Caretta caretta papillomavirus 1". Virology. 383 (1): 131–5. January 2009. doi:10.1016/j.virol.2008.09.022. PMID 18973915.
- "Detection of herpesvirus-like and papillomavirus-like particles associated with diseases of tortoises". The Veterinary Record. 143 (23): 639. December 1998. PMID 9881444.
- "Novel snake papillomavirus does not cluster with other non-mammalian papillomaviruses". Virology Journal. 8: 436. September 2011. doi:10.1186/1743-422X-8-436. PMID 21910860.
- "Chapter 1: HPV in the etiology of human cancer". Vaccine. 24 Suppl 3 (3): S3/1–10. August 2006. doi:10.1016/j.vaccine.2006.05.115. PMID 16949995.
- "Cutaneous and mucosal human papillomaviruses differ in net surface charge, potential impact on tropism". Virology Journal. 5: 118. October 2008. doi:10.1186/1743-422X-5-118. PMID 18854037.
- "The papillomavirus life cycle". Journal of Clinical Virology. 32 Suppl 1: S7–15. March 2005. doi:10.1016/j.jcv.2004.12.006. PMID 15753007.
- "Complete Genome Sequence of a Novel Human Betapapillomavirus, HPV-159". Genome Announcements. 1 (3): e00298–13. May 2013. doi:10.1128/genomeA.00298-13. PMID 23723399.
- "Virus Taxonomy: 2018b Release". International Committee on Taxonomy of Viruses. February 2019. مؤرشف من الأصل في 16 مايو 202026 مارس 2019.
- "Virus Taxonomy: 2014 Release". ICTV. مؤرشف من الأصل في 10 فبراير 201715 يونيو 2015.
- "Classification of papillomaviruses (PVs) based on 189 PV types and proposal of taxonomic amendments". Virology. 401 (1): 70–9. May 2010. doi:10.1016/j.virol.2010.02.002. PMID 20206957.
- Campo MS, المحرر (2006). Papillomavirus Research: From Natural History To Vaccines and Beyond. Caister Academic Press. . [1]. مؤرشف من الأصل في 22 مايو 2016.
- "Animal papillomaviruses". Virology. 445 (1–2): 213–23. October 2013. doi:10.1016/j.virol.2013.05.007. PMID 23711385.
- "Concurrence of Iridovirus, Polyomavirus, and a Unique Member of a New Group of Fish Papillomaviruses in Lymphocystis Disease-Affected Gilthead Sea Bream". Journal of Virology. 90 (19): 8768–79. October 2016. doi:10.1128/JVI.01369-16. PMID 27440877.
- "Analysis of the genetic diversity and phylogenetic relationships of putative human papillomavirus types". The Journal of General Virology. 94 (Pt 11): 2480–8. November 2013. doi:10.1099/vir.0.055137-0. PMID 23997181.
- "Healthy skin of many animal species harbors papillomaviruses which are closely related to their human counterparts". Journal of Virology. 76 (24): 12537–42. December 2002. doi:10.1128/JVI.76.24.12537-12542.2002. PMID 12438579.
- Holliday, Chuck. "Prof. Chuck Holliday's www page at Lafayette College » Jackalopes". مؤرشف من الأصل في 18 يوليو 201413 يوليو 2014.
- "Cottontail rabbit papillomavirus (CRPV) model system to test antiviral and immunotherapeutic strategies". Antiviral Chemistry & Chemotherapy. 16 (6): 355–62. 2005. doi:10.1177/095632020501600602. PMID 16331841.
- "Sequence homologies between bovine papillomavirus genomes mapped by a novel low-stringency heteroduplex method". Virology. 143 (2): 603–11. June 1985. doi:10.1016/0042-6822(85)90398-8. PMID 2998027.
- "Primary structure of the E6 protein of Micromys minutus papillomavirus and Mastomys natalensis papillomavirus". Nucleic Acids Research. 20 (11): 2889. June 1992. doi:10.1093/nar/20.11.2889. PMID 1319576.
- "A novel papillomavirus in Adélie penguin (Pygoscelis adeliae) faeces sampled at the Cape Crozier colony, Antarctica". The Journal of General Virology. 95 (Pt 6): 1352–65. June 2014. doi:10.1099/vir.0.064436-0. PMID 24686913. مؤرشف من الأصل في 16 مايو 2020.
- "Quantifying the phylodynamic forces driving papillomavirus evolution". Molecular Biology and Evolution. 28 (7): 2101–13. July 2011. doi:10.1093/molbev/msr030. PMID 21285031.
- "The genetic drift of human papillomavirus type 16 is a means of reconstructing prehistoric viral spread and the movement of ancient human populations". Journal of Virology. 67 (11): 6413–23. November 1993. doi:10.1128/JVI.67.11.6413-6423.1993. PMID 8411343.
- "Worldwide genomic diversity of the high-risk human papillomavirus types 31, 35, 52, and 58, four close relatives of human papillomavirus type 16". Journal of Virology. 79 (21): 13630–40. November 2005. doi:10.1128/JVI.79.21.13630-13640.2005. PMID 16227283.
- "Human papillomavirus type 13 and pygmy chimpanzee papillomavirus type 1: comparison of the genome organizations". Virology. 190 (2): 587–96. October 1992. doi:10.1016/0042-6822(92)90896-W. PMID 1325697.
- Willemsen A, Bravo IG (2019) Origin and evolution of papillomavirus (onco)genes and genomes. Philos Trans R Soc Lond B Biol Sci. 374(1773):20180303
- Murahwa AT, Nindo F, Onywera H, Meiring TL, Martin DP, Williamson AL (2019) Evolutionary dynamics of ten novel Gamma-PVs: insights from phylogenetic incongruence, recombination and phylodynamic analyses. BMC Genomics 20(1):368
- "Polyoma virus capsid structure at 22.5 A resolution". Nature. 295 (5845): 110–5. January 1982. doi:10.1038/295110a0. PMID 6276752.
- "A tiling approach to virus capsid assembly explaining a structural puzzle in virology". Journal of Theoretical Biology. 226 (4): 477–82. February 2004. doi:10.1016/j.jtbi.2003.10.006. PMID 14759653.
- "The L1 major capsid protein of human papillomavirus type 11 recombinant virus-like particles interacts with heparin and cell-surface glycosaminoglycans on human keratinocytes". The Journal of Biological Chemistry. 274 (9): 5810–22. February 1999. doi:10.1074/jbc.274.9.5810. PMID 10026203.
- "Human papillomavirus infection requires cell surface heparan sulfate". Journal of Virology. 75 (3): 1565–70. February 2001. doi:10.1128/JVI.75.3.1565-1570.2001. PMID 11152531.
- "Identification of the alpha6 integrin as a candidate receptor for papillomaviruses". Journal of Virology. 71 (3): 2449–56. March 1997. doi:10.1128/JVI.71.3.2449-2456.1997. PMID 9032382.
- "Expression of the alpha6 integrin confers papillomavirus binding upon receptor-negative B-cells". Virology. 261 (2): 271–9. September 1999. doi:10.1006/viro.1999.9825. PMID 10497112.
- "Analysis of the infectious entry pathway of human papillomavirus type 33 pseudovirions". Virology. 299 (2): 279–287. August 2002. doi:10.1006/viro.2001.1493. PMID 12202231.
- "Papillomaviruses infect cells via a clathrin-dependent pathway". Virology. 307 (1): 1–11. March 2003. doi:10.1016/S0042-6822(02)00143-5. PMID 12667809.
- "A membrane-destabilizing peptide in capsid protein L2 is required for egress of papillomavirus genomes from endosomes". Journal of Virology. 80 (2): 759–68. January 2006. doi:10.1128/JVI.80.2.759-768.2006. PMID 16378978.
- "Establishment of papillomavirus infection is enhanced by promyelocytic leukemia protein (PML) expression". Proceedings of the National Academy of Sciences of the United States of America. 101 (39): 14252–7. September 2004. Bibcode:2004PNAS..10114252D. doi:10.1073/pnas.0404229101. PMID 15383670.
- Zhang, Pengwei; Monteiro Da Silva, Gabriel; Deatherage, Catherine; Burd, Christopher; Dimaio, Daniel (2018). "Cell-Penetrating Peptide Mediates Intracellular Membrane Passage of Human Papillomavirus L2 Protein to Trigger Retrograde Trafficking". Cell. 174 (6): 1465–1476.e13. doi:10.1016/j.cell.2018.07.031. PMID 30122350.
- "The etiologic role of human papillomavirus in penile cancers: a study in Vietnam". British Journal of Cancer. 108 (1): 229–33. January 2013. doi:10.1038/bjc.2012.583. PMID 23299525.
- "The role of human papillomavirus in oral squamous cell carcinoma (review)". Georgian Medical News (213): 32–6. December 2012. PMID 23293230.
- "Role of Human Papillomavirus Infection in the Etiology of Vulvar Cancer in Italian Women". Infect Agent Cancer. 20. 2020. doi:10.1186/s13027-020-00286-8. PMID 32266002. مؤرشف من الأصل في 12 أبريل 2020.
- "Human papillomaviruses in urological malignancies: a critical assessment". Urologic Oncology. 32 (1): 46.e19–27. January 2014. doi:10.1016/j.urolonc.2013.06.012. PMID 24140249.
- "PDB101: Molecule of the Month: Human Papillomavirus and Vaccines". RCSB: PDB-101. مؤرشف من الأصل في 17 أكتوبر 201814 مايو 2018.
- Guan, J; Bywaters, S.M; Brendle, S.A; Ashley, R.E; Makhov, A.M; Conway, J.F; Christensen, N.D; Hafenstein, S (2018). "High-Resolution Structure Analysis of Antibody V5 Conformational Epitope on Human Papillomavirus 16". doi:10.2210/pdb6bt3/pdb.
- "Biosynthesis of human papillomavirus from a continuous cell line upon epithelial differentiation". Science. 257 (5072): 971–3. August 1992. Bibcode:1992Sci...257..971M. doi:10.1126/science.1323879. PMID 1323879.
- "Propagation, infection, and neutralization of authentic HPV16 virus". Virology. 322 (2): 213–9. May 2004. doi:10.1016/j.virol.2004.02.011. PMID 15110519.
- Goodsell, D.S (2018). "Human Papillomavirus and Vaccines". RCSB Protein Data Bank. doi:10.2210/rcsb_pdb/mom_2018_5.
- "Papillomavirus genome structure, expression, and post-transcriptional regulation". Frontiers in Bioscience. 11: 2286–302. September 2006. doi:10.2741/1971. PMID 16720315.
- "The E7 oncoprotein is translated from spliced E6*I transcripts in high-risk human papillomavirus type 16- or type 18-positive cervical cancer cell lines via translation reinitiation". Journal of Virology. 80 (9): 4249–63. May 2006. doi:10.1128/JVI.80.9.4249-4263.2006. PMID 16611884.
- "Construction of a full transcription map of human papillomavirus type 18 during productive viral infection". Journal of Virology. 85 (16): 8080–92. August 2011. doi:10.1128/JVI.00670-11. PMID 21680515.
- "Control of the papillomavirus early-to-late switch by differentially expressed SRp20". Journal of Virology. 83 (1): 167–80. January 2009. doi:10.1128/JVI.01719-08. PMID 18945760.
- "Brd4: tethering, segregation and beyond". Trends in Microbiology. 12 (12): 527–9. December 2004. doi:10.1016/j.tim.2004.10.002. PMID 15539109.
- "Identification of the human papilloma virus-1a E4 gene products". The EMBO Journal. 5 (2): 355–62. February 1986. doi:10.1002/j.1460-2075.1986.tb04219.x. PMID 3011404.
- "Mucosal human papillomaviruses encode four different E5 proteins whose chemistry and phylogeny correlate with malignant or benign growth". Journal of Virology. 78 (24): 13613–26. December 2004. doi:10.1128/JVI.78.24.13613-13626.2004. PMID 15564472.
- PDB 2I0I - تصفح: نسخة محفوظة 2018-06-28 على موقع واي باك مشين.
- "The human papillomavirus type 11 and 16 E6 proteins modulate the cell-cycle regulator and transcription cofactor TRIP-Br1". Virology. 317 (1): 155–64. December 2003. doi:10.1016/j.virol.2003.08.008. PMID 14675634.
- "Interactions of the PDZ-protein MAGI-1 with adenovirus E4-ORF1 and high-risk papillomavirus E6 oncoproteins". Oncogene. 19 (46): 5270–80. November 2000. doi:10.1038/sj.onc.1203906. PMID 11077444.
- "iHOP information Hyperlinked over Proteins UBE3A". مؤرشف من الأصل في 27 سبتمبر 200701 مايو 2007.
- "Biochemistry, Nottingham University – 3.0 Enzymes of the Ubiquitin Pathway". مؤرشف من الأصل في 06 مايو 200701 مايو 2007.
- "The global transcriptional effects of the human papillomavirus E6 protein in cervical carcinoma cell lines are mediated by the E6AP ubiquitin ligase". Journal of Virology. 79 (6): 3737–47. March 2005. doi:10.1128/JVI.79.6.3737-3747.2005. PMID 15731267.
- "Binding of high-risk human papillomavirus E6 oncoproteins to the human homologue of the Drosophila discs large tumor suppressor protein". Proceedings of the National Academy of Sciences of the United States of America. 94 (21): 11612–6. October 1997. Bibcode:1997PNAS...9411612K. doi:10.1073/pnas.94.21.11612. PMID 9326658.
- "Requirement of E7 oncoprotein for viability of HeLa cells". Microbes and Infection. 8 (4): 984–93. April 2006. doi:10.1016/j.micinf.2005.10.015. PMID 16500131.
- "The B subgroup bovine papillomaviruses lack an identifiable E6 open reading frame". Molecular Carcinogenesis. 4 (5): 382–7. 1991. doi:10.1002/mc.2940040510. PMID 1654923.
- "Identification of human papillomavirus type 16 L1 surface loops required for neutralization by human sera". Journal of Virology. 80 (10): 4664–72. May 2006. doi:10.1128/JVI.80.10.4664-4672.2006. PMID 16641259.
- Papavasiliou, Nina, المحرر (2009). "Two highly conserved cysteine residues in HPV16 L2 form an intramolecular disulfide bond and are critical for infectivity in human keratinocytes". PLOS One. 4 (2): e4463. Bibcode:2009PLoSO...4.4463C. doi:10.1371/journal.pone.0004463. PMID 19214230.
- "Cleavage of the papillomavirus minor capsid protein, L2, at a furin consensus site is necessary for infection". Proceedings of the National Academy of Sciences of the United States of America. 103 (5): 1522–7. January 2006. Bibcode:2006PNAS..103.1522R. doi:10.1073/pnas.0508815103. PMID 16432208.
- "Interaction of L2 with beta-actin directs intracellular transport of papillomavirus and infection". The Journal of Biological Chemistry. 278 (14): 12546–53. April 2003. doi:10.1074/jbc.M208691200. PMID 12560332.
- "Interaction of tSNARE syntaxin 18 with the papillomavirus minor capsid protein mediates infection". Journal of Virology. 79 (11): 6723–31. June 2005. doi:10.1128/JVI.79.11.6723-6731.2005. PMID 15890910.
- "Cross-neutralization of cutaneous and mucosal Papillomavirus types with anti-sera to the amino terminus of L2". Virology. 337 (2): 365–72. July 2005. doi:10.1016/j.virol.2005.04.011. PMID 15885736.