
En microbiologie et en biologie moléculaire, un plasmide est une molécule d'ADN distincte de l'ADN chromosomique, capable de réplication autonome et non essentielle à la survie de la cellule. Les plasmides sont bicaténaires (constitués de deux brins complémentaires) et généralement circulaires. On les trouve presque exclusivement dans les bactéries et parfois dans d'autres micro-organismes comme le plasmide 2Mu que l'on trouve dans la levure Saccharomyces cerevisiae (levure de boulangerie), un eucaryote unicellulaire. Les plasmides sont présents en nombre de copies variable : les grands plasmides naturels (>50 kb) sont souvent présents à une ou deux copies par cellule, comme le chromosome. Les plasmides construits par génie génétique pour des utilisations biotechnologiques (expression de protéine recombinante, clonage de gène, séquençage de l'ADN) ont souvent été modifiés pour être présents à un nombre de copies beaucoup plus élevé, de l'ordre quelques dizaines à quelques centaines, ce qui permet une amplification de la production d'ADN ou de protéines.
Des plasmides différents peuvent coexister dans une cellule (sous condition de leur compatibilité mutuelle). Certains plasmides sont capables de s'intégrer aux chromosomes ; on appelle ces plasmides des épisomes.
Les plasmides participent aux transferts horizontaux de gènes, donc entre espèces et populations différentes. Ils concourent à la dissémination de gènes conférant des avantages sélectifs (par exemple des résistances aux antibiotiques, à des biocides ou encore des facteurs de virulence). La mobilité des plasmides (par conjugaison) au sein des populations bactériennes accroît le spectre d'hôte des gènes impliqués dans la virulence. Ces gènes offrent en contrepartie un avantage sélectif pour le plasmide et les bactéries hôtes. On conçoit donc la nature quasi-ubiquitaire et persistante des plasmides chez les bactéries pathogènes. Les bactéries stressées transfèrent plus facilement ainsi leurs capacités de résistance à des populations bactériennes distantes, éventuellement géographiquement éloignées, ce qui explique la propagation à grande échelle de gènes de résistance aux antibiotiques ou à des biocides largement dispersés par l'homme dans l'environnement[1].
Histoire scientifique
Le terme plasmide fut introduit par le biologiste moléculaire américain Joshua Lederberg en 1952[2]. Un plasmide porte un ensemble de gènes permettant sa propre réplication, et fréquemment des gènes d'antibiorésistance qui apportent un avantage aux bactéries qui les contiennent.
Réplication et transmission

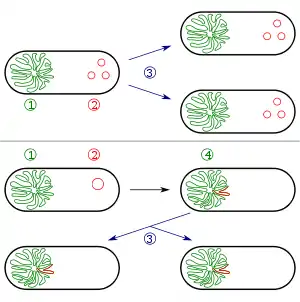
1 et 2. Gène(s) codant la (les) résistance(s).
3. Origine de réplication.
Chaque plasmide contient au moins une séquence d'ADN qui sert d'origine de réplication, ou ori (un point de départ de réplication de l'ADN), permettant à l'ADN plasmidique d'être dupliqué indépendamment du chromosome bactérien ou de levure, en utilisant la « machinerie » de la cellule hôte. Les plasmides peuvent être circulaires, ou parfois linéaires, présentant une ressemblance superficielle avec les chromosomes eucaryotes.
Comme les plasmides présents chez les bactéries ne portent habituellement pas de gènes essentiels à la cellule procaryote, leur pérennité dans une lignée de bactéries dépend donc de divers moyens de stabilisation des plasmides, laquelle résulte de divers processus de sélection et de conditions environnementales. De plus, les plasmides peuvent servir de synthétiseur pour les bactéries.
Les plasmides peuvent se transmettre d'une bactérie à une autre grâce à la conjugaison bactérienne par l'intermédiaire de pili sexuels. Lors de la division cellulaire, les plasmides se répartissent dans les cellules filles de façon totalement aléatoire (contrairement aux chromosomes) ainsi, même si la probabilité reste faible, il se peut qu'une des deux cellules filles ne possède aucun plasmide. La probabilité augmente avec la diminution du nombre de plasmides présents dans la cellule mère.
Épisomes
1. ADN chromosomique.
2. Plasmides.
3. Division cellulaire.
4. ADN chromosomique avec plasmides intégrés.
Un épisome est un plasmide qui peut s'intégrer dans l'ADN chromosomique de la cellule hôte. De ce fait, il peut rester intact pendant de longues périodes, être dupliqué à chaque division cellulaire de l'hôte, et devenir partie intégrante de son patrimoine génétique. Le terme n'est plus en usage pour les plasmides, depuis qu'il a été établi qu'une région d'homologie avec le chromosome, comme un transposon, fait d'un plasmide un épisome. Dans les systèmes mammifères, le terme épisome fait référence à un ADN circulaire (comme un génome viral) attaché au chromosome de la cellule-hôte de façon non-covalente.
Vecteurs
Les vecteurs sont des plasmides issus de constructions génétiques faites en laboratoire et présentant un certain nombre de caractéristiques pour des applications en thérapie génique et en biotechnologie. Ils contiennent en général une région contenant des sites d'enzymes de restriction permettant d'insérer des fragments d'ADN, pour le clonage, le séquençage ou l'expression de protéine. Ils contiennent également un gène permettant de sélectionner les bactéries contenant le plasmide, en général un gène de résistance à un antibiotique. Ils possèdent également parfois des gènes rapporteurs, permettant la purification ou la détection des protéines recombinantes : protéine verte fluorescente, glutathion-S-transférase, étiquette poly-histidine.
Les vecteurs sont des outils essentiels de la biologie moléculaire.
Propriétés codées par les plasmides
Plasmides conjugatifs
Les plasmides conjugatifs sont les premiers plasmides qui ont été découverts chez la bactérie Escherichia coli dans les années 1950. On les appelle aussi facteurs de fertilité ou plasmides F. Ces plasmides confèrent à la bactérie hôte la capacité de synthèse de pili dit sexuels. Par l'intermédiaire de ces pili, la bactérie porteuse (donneuse) peut transférer une copie du plasmide F par processus de conjugaison bactérienne. Les plasmides F possèdent au minimum une origine de réplication et tous les gènes nécessaires à la synthèse des pili et du transfert du plasmide. Certains plasmides F sont des épisomes, c'est-à-dire qu'ils peuvent s'intégrer dans le génome chromosomique.
Plasmides de résistance
Les plasmides de résistance, appelés aussi plasmides ou facteurs R, codent des résistances aux antibiotiques et aux métaux lourds.
En 1959, au Japon, on a retrouvé chez les malades atteints de dysenterie bactérienne une insensibilité à tout traitement antibiotique. En fait, la bactérie responsable, Shigella dysenteriae, portait des gènes de résistance à plusieurs antibiotiques encore jamais rencontrés. Par la suite, on en a retrouvé chez d'autres bactéries (comme E. coli) et ces plasmides furent baptisés R Factor pour facteurs de résistance (R factors que l'on considérait comme des éléments constitués de RTF Factors ou resistance transfer factors. Ces descriptions réalisées par des équipes japonaises (et publiées en japonais) ont été synthétisées dans un article écrit par Watanabe en 1963[3] ayant fait date dans l'histoire de la microbiologie, puisqu'il ouvrit la voie à la découverte des plasmides dont la première description fut réalisée par Joshua Lederberg et coll.
Ces plasmides peuvent protéger la cellule par différents moyens que sont la modification de cible (la cible de l'action d'un antibiotique se voit modifiée, rendant la bactérie résistante à cet antibiotique), la résistance enzymatique (la bactérie produit une substance -une enzyme- capable d'inactiver directement l'antibiotique en la dénaturant, l'hydrolisant etc. (c'est le cas par exemple des bêta-lactamases), imperméabilité de l'enveloppe cellulaire (par modification des porines) et efflux actif (lié à des pompes d'efflux) -ces deux mécanismes ayant pour effet d'empêcher la pénétration intra-cellulaire de l'antibiotique ou de favoriser son extrusion active hors du micro-organisme).
Plasmides métaboliques
Les plasmides métaboliques portent des gènes permettant l'utilisation de certains nutriments. Chez E. coli, les gènes portés par ces plasmides sont par exemple : l'utilisation du citrate comme source de carbone, la production de soufre, l'hydrolyse de l'urée. Chez les salmonelles on a observé la dégradation du lactose ce qui est totalement inhabituel chez ce genre bactérien. La plupart de ces plasmides codent la synthèse d'une ou de plusieurs enzymes.
Plasmides de virulence
Il s'avère que les bactéries pathogènes hébergent très souvent des plasmides conjugatifs qui participent à la pathogénicité. Les plasmides de virulence portent des gènes codant des facteurs de virulence, ayant un rôle dans le pouvoir pathogène des bactéries. Par exemple les Escherichia coli entérotoxigéniques (ETEC) responsable de la diarrhée du voyageur (ou tourista) hébergent au moins deux plasmides, l'un portant les gènes codant un facteur de colonisation, l'autre codant des toxines.
De même, les déterminants du pouvoir invasif des Shigella sont portés par un plasmide (pInv). Chez d'autres bactéries pathogènes (par exemple Salmonella), ces plasmides codent un complexe protéique situé sur la paroi de la bactérie : c'est le complexe pili-adhésine qui permet à la bactérie d'adhérer sur des récepteurs hydrocarbonés situés à la surface de certaines cellules eucaryotes notamment les entérocytes. Certains plasmides codent des facteurs tumorigènes ; c'est notamment les cas pour la « galle du collet » due à un plasmide Ti (pour tumor inducing) hébergé par les bactéries du genre Agrobacterium.
Plasmides de bactériocines et de résistance aux biocides ou à des polluants
Ces plasmides codent la synthèse d'une protéine extracellulaire dont la biosynthèse est létale pour la bactérie productrice ainsi que pour les autres bactéries non-productrices environnantes. Cependant, ces plasmides codent aussi une deuxième protéine intracellulaire de résistance à cette première toxine. Les bactériocines agissent sur des fonctions vitales de la bactérie.
Chez E. coli, on trouvera différentes catégories de bactériocines (colicines codées par les plasmides col) et par exemple le gène colE1 code une endonucléase et le gène colE3, une ribonucléase qui inactive les ribosomes.
Chez des bactéries Roseobacter, dont la plasticité génétique est reconnue et qui jouent un rôle importa dans le cycle du carbone et du soufre, des chercheurs ont trouvé un plasmide qui est aussi retrouvé dans plusieurs autres genres de bactéries, en des lieux géographiquement éloignés, révélant des "événements récents de transfert de gènes horizontaux à l'échelle mondiale"[1]. Ce plasmide contient des gènes de détoxification de polluants anthropiques ; tout comme les plasmides porteurs de résistance aux antibiotiques, il semble résulter d'une sélection pour les activités humaines[1]
Fabrication de plasmides recombinants
Un plasmide recombinant est un plasmide dans lequel a été inséré un fragment d'ADN étranger. Exemple: Lors du séquençage de l'ADN (voir ci-dessous),les plasmides accueillant de L'ADN génomique sont appelés plasmides recombinants.
Application en génie génétique
Application pour la production de molécule
On désire faire produire par des bactéries une certaine protéine (protéine d'intérêt). C'est par l'intermédiaire de plasmides qu'on introduira dans des cultures de bactéries un gène codant notre protéine d'intérêt et un gène de résistance à un antibiotique X. On sélectionnera les bactéries ayant intégré les plasmides en faisant pousser les colonies bactériennes sur un milieu contenant l'antibiotique X; les bactéries n'ayant pas intégré le plasmide ne se développeront pas. Cette technique est déjà grandement utilisée pour la production de somatostatine, hormone de croissance, humaine. Avant la connaissance de cette méthode, l'hormone était récupérée sur les morts, engendrant de nombreux problèmes de transmission de pathologies non repérées sur les cadavres prélevés. Depuis que la production de celle-ci est effectuée par des bactéries modifiées génétiquement via les plasmides, les patients souffrant d'un déficit d'hormone de croissance peuvent bénéficier de ces protéines qui leur manquent sans ce risque de transmission inter-humaine. Des tests sont également en cours pour la production d'un médicament contre les troubles liés à la mucoviscidose, maladie génétique pour laquelle la biomédecine manque pour le moment de soins efficaces.
Il est également possible de produire des protéines d'intérêt par d'autres types d'hôtes hétérologues comme des cultures eucaryotes ou mammifères en particulier. Cela présente l'avantage de disposer de la machinerie de modifications post-traductionnelles telles que la glycosylation, absente chez les procaryotes. Or pour la plupart des protéines relativement complexes, celles-ci ne deviennent bioactives qu'une fois ces modifications opérées. Il est possible d'effectuer ces modifications à l'issue d'une production par une culture procaryote, mais cela reste encore fort complexe et exigeant.
Application pour le séquençage
Cette application est particulièrement utile en génie génétique pour le séquençage d'ADN : l'ADN d'une cellule quelconque est difficile à séquencer car il dépasse très souvent 400 000 kb (1 kilobase = 1 000 bases) et se trouve en faible quantité. Ainsi, pour rendre le séquençage plus facile, on découpe l'ADN à analyser et l'ADN plasmidique avec une enzyme de restriction, dans des conditions spécifiques ; l'ADN à séquencer va s'intégrer dans l'ADN plasmidique. Le plasmide recombinant sera transféré dans le hyaloplasme bactérien. Une fois mise en culture, la bactérie va répliquer l'ADN plasmidique (et donc le fragment à séquencer) en grande quantité. Après avoir extrait l'ADN à séquencer, et éliminé le reste d'ADN plasmidique par des enzymes de restriction, on récupère les fragments d'ADN reproduits à plusieurs milliers d'exemplaires, que l'on peut alors séquencer facilement.
Notes et références
- 1 2 3 Petersen, J., Vollmers, J., Ringel, V., Brinkmann, H., Ellebrandt-Sperling, C., Spröer, C., ... & Kaster, A. K. (2019). A marine plasmid hitchhiking vast phylogenetic and geographic distances. Proceedings of the National Academy of Sciences, 116(41), 20568-20573.
- ↑ Lederberg J (1952), Physiol. Rev. 32, 403-430.
- ↑ Watanabe T., Infective heredity of multiple drug resistance in Bacteria. Bacteriol Rev 1963;27:87-115