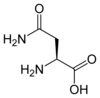
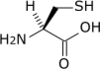
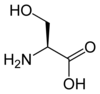
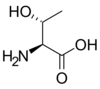
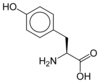
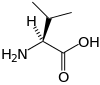
-N-Formylmethionine_V.1.svg.png.webp)
Elle amorce la biosynthèse des protéines chez les procaryotes et dans les mitochondries et les chloroplastes des eucaryotes, mais pas dans le cytosol de ces derniers.
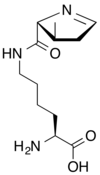
Elle dérive de la méthionine par adjonction d'un groupe aldéhyde sur l'atome d'azote de l'amine primaire
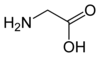
Un acide aminé protéinogène est un acide aminé incorporé dans les protéines lors de la traduction de l'ARN messager par les ribosomes[1]. Il existe en tout 22 acides aminés protéinogènes, ce qualificatif étant forgé à partir de racines grecques signifiant précisément « qui donne naissance aux protéines ». Parmi ceux-ci, 20 acides aminés, dits acides aminés standards, sont codés directement par un codon de l'ADN nucléaire. Deux autres acides aminés protéinogènes sont codés indirectement par des codons-stop qui sont recodés en codons d'acides aminés en présence de séquences d'insertion particulières, appelées élément SECIS pour la sélénocystéine et élément PYLIS pour la pyrrolysine. La pyrrolysine n'est présente que dans les protéines de certaines archées méthanogènes, de sorte que les eucaryotes et les bactéries n'utilisent que 21 acides aminés protéinogènes.
Bien qu'elle présente une chaîne latérale identique à celle de la méthionine, la N-formylméthionine est parfois considérée comme un 23e acide aminé protéinogène car, chez les procaryotes ainsi que dans les mitochondries et les chloroplastes des eucaryotes, c'est elle qui est incorporée par les ribosomes à la place de la méthionine au début des chaînes polypeptidiques lors de la traduction de l'ARN messager ; ce résidu est souvent éliminé par la suite par modification post-traductionnelle de la protéine néoformée[2].

Le corps humain est capable de synthétiser 12 des 21 acides aminés protéinogènes qu'il utilise, tandis que neuf d'entre eux, dits acides aminés essentiels, doivent lui être apportés par l'alimentation : histidine, isoleucine, leucine, lysine, méthionine, phénylalanine, thréonine, tryptophane et valine.
En revanche, les acides aminés non protéinogènes peuvent être incorporés dans les protéines au cours de modifications post-traductionnelles, comme le γ-carboxyglutamate ou l'hydroxyproline, voire être totalement absents des protéines, comme l'acide γ-aminobutyrique ou la DOPA.
Structure des acides aminés protéinogènes
La planche ci-dessous présente la structure non ionisée des 22 acides aminés protéinogènes ; en solution aqueuse à pH physiologique, les fonctions carboxyle –COOH et amine –NH2 sont ionisées en carboxylate –COO− et ammonium –NH3+, les espèces chimiques correspondantes sont des zwitterions.
| |||||||||||||||||||||||||||||||||||||||||||||||||
| Structure des 22 acides aminés protéinogènes. La pyrrolysine et la sélénocystéine (ci-dessus grisées) sont spécifiques à certaines protéines : - la pyrrolysine ne se rencontre que chez certaines archées méthanogènes, - la sélénocystéine est présente également chez les eucaryotes mais a priori dans quelques dizaines d'enzymes de la famille des oxydoréductases. Les 20 autres acides aminés, dits standards, sont en revanche universellement distribués chez tous les êtres vivants connus. |
Propriétés chimiques
Le tableau ci-dessous résume les principales propriétés chimiques des acides aminés protéinogènes et de leur chaîne latérale. Les valeurs proposées pour le point isoélectrique et les constantes d'acidité sont celles de l'université de Calgary[3], en Alberta.
| Acide aminé | Abrév. | Masse (Da) | pI | Rayon de van der Waals |
pKa1 (α-COOH) |
pKa2 (α-+NH3) |
Chaîne latérale | |||
|---|---|---|---|---|---|---|---|---|---|---|
| pKR | Structure | Nature | ||||||||
| Alanine | A | Ala | 89,09404 | 6,00 | 67 | 2,34 | 9,60 | –CH3 | Hydrophobe | |
| Arginine | R | Arg | 174,20274 | 10,76 | 148 | 2,17 | 9,04 | 12,48 | –CH2CH2CH2–NHC(=NH)NH2 | Polaire, très basique |
| Asparagine | N | Asn | 132,11904 | 5,41 | 96 | 2,02 | 8,80 | –CH2CONH2 | Polaire | |
| Aspartate | D | Asp | 133,10384 | 2,77 | 91 | 1,88 | 9,60 | 3,65 | –CH2COOH | Polaire, acide |
| Cystéine | C | Cys | 121,15404 | 5,07 | 86 | 1,96 | 10,70 | 8,33 | –CH2SH | Hydrophobe[4], faiblement acide |
| Glutamate | E | Glu | 147,13074 | 3,22 | 109 | 2,19 | 9,67 | 4,25 | –CH2CH2COOH | Polaire, acide |
| Glutamine | Q | Gln | 146,14594 | 5,65 | 114 | 2,17 | 9,13 | –CH2CH2CONH2 | Polaire | |
| Glycine | G | Gly | 75,06714 | 5,97 | 48 | 2,34 | 9,60 | –H | Hydrophobe | |
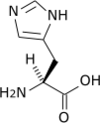
| Histidine | H | His | 155,15634 | 7,59 | 118 | 1,82 | 9,17 | 6,00 | –CH2-C3H3N2 | Polaire, faiblement basique, aromatique |
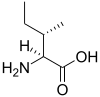
| Isoleucine | I | Ile | 131,17464 | 6,02 | 124 | 2,36 | 9,60 | –CH(CH3)CH2CH3 | Hydrophobe, aliphatique | |
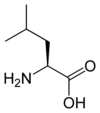
| Leucine | L | Leu | 131,17464 | 5,98 | 124 | 2,36 | 9,60 | –CH2CH(CH3)2 | Hydrophobe, aliphatique | |
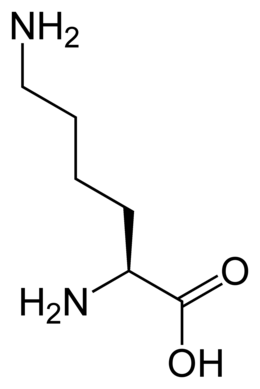
| Lysine | K | Lys | 146,18934 | 9,74 | 135 | 2,18 | 8,95 | 10,53 | –CH2CH2CH2CH2NH2 | Polaire, basique |
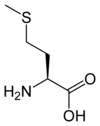
| Méthionine | M | Met | 149,20784 | 5,74 | 124 | 2,28 | 9,21 | –CH2CH2–S–CH3 | Hydrophobe | |
| Phénylalanine | F | Phe | 165,19184 | 5,48 | 135 | 1,83 | 9,13 | –CH2–C6H5 | Hydrophobe, aromatique | |
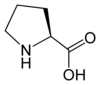
| Proline | P | Pro | 115,13194 | 6,30 | 90 | 1,99 | 10,60 | –CH2CH2CH2- | Hydrophobe | |
| Pyrrolysine | O | Pyl | 255,31 | –CH2CH2CH2CH2NHCO–C4H5N–CH3 | Polaire, faiblement basique | |||||
| Sélénocystéine | U | Sec | 168,053 | 5,47 | 5,73 | –CH2SeH | ||||
| Sérine | S | Ser | 105,09344 | 5,68 | 73 | 2,21 | 9,15 | 5,68 | –CH2OH | Polaire, faiblement acide |
| Thréonine | T | Thr | 119,12034 | 5,60 | 93 | 2,09 | 9,10 | 5,53 | –CH(OH)CH3 | Polaire, faiblement acide |
| Tryptophane | W | Trp | 204,22844 | 5,89 | 163 | 2,83 | 9,39 | 5,885 | –CH2–C8H6N | Polaire, faiblement basique, aromatique |
| Tyrosine | Y | Tyr | 181,19124 | 5,66 | 141 | 2,20 | 9,11 | 10,07 | –CH2–C6H4OH | Polaire, faiblement acide, aromatique |
| Valine | V | Val | 117,14784 | 5,96 | 105 | 2,32 | 9,62 | –CH(CH3)2 | Hydrophobe, aliphatique | |
La valeur du pKR ici donnée correspond à l'acide aminé libre. Cette valeur varie sensiblement pour les résidus d'acides aminés présents dans les protéines, comme l'indique le tableau ci-dessous :
Expression génétique
Le tableau ci-dessous présente les codons d'ARN messager correspondant à chacun des 22 acides aminés protéinogènes. Les valeurs relatives à l'abondance relative des acides aminés dans les protéines varient légèrement suivant les espèces et les bases de données utilisées. Elles sont données ici à titre indicatif.
| Acide aminé | Abrév. | Codon(s) d'ARN messager | Occurrence dans les protéines[5] |
Occurrence chez les vertébrés[6] |
Essentiel chez l'homme | |
|---|---|---|---|---|---|---|
| Alanine | A | Ala | GCU, GCC, GCA, GCG | 9,0 % | 7,4 % | Non |
| Arginine | R | Arg | CGU, CGC, CGA, CGG, AGA, AGG | 4,7 % | 4,2 % | Selon les cas |
| Asparagine | N | Asn | AAU, AAC | 4,4 % | 4,4 % | Non |
| Aspartate | D | Asp | GAU, GAC | 5,5 % | 5,9 % | Non |
| Cystéine | C | Cys | UGU, UGC | 2,8 % | 3,3 % | Selon les cas |
| Glutamate | E | Glu | GAA, GAG | 6,2 % | 5,8 % | Selon les cas |
| Glutamine | Q | Gln | CAA, CAG | 3,9 % | 3,7 % | Non |
| Glycine | G | Gly | GGU, GGC, GGA, GGG | 7,5 % | 7,4 % | Non |
| Histidine | H | His | CAU, CAC | 2,1 % | 2,9 % | Oui |
| Isoleucine | I | Ile | AUU, AUC, AUA | 4,6 % | 3,8 % | Oui |
| Leucine | L | Leu | UUA, UUG, CUU, CUC, CUA, CUG | 7,5 % | 7,6 % | Oui |
| Lysine | K | Lys | AAA, AAG | 7,0 % | 7,2 % | Oui |
| Méthionine | M | Met | AUG | 1,7 % | 1,8 % | Oui |
| Phénylalanine | F | Phe | UUU, UUC | 3,5 % | 4,0 % | Oui |
| Proline | P | Pro | CCU, CCC, CCA, CCG | 4,6 % | 5,0 % | Non |
| Pyrrolysine | O | Pyl | UAG avec élément PYLIS | — | — | — |
| Sélénocystéine | U | Sel | UGA avec élément SECIS | — | — | Non |
| Sérine | S | Ser | UCU, UCC, UCA, UCG, AGU, AGC | 7,1 % | 8,1 % | Non |
| Thréonine | T | Thr | ACU, ACC, ACA, ACG | 6,0 % | 6,2 % | Oui |
| Tryptophane | W | Trp | UGG | 1,1 % | 1,3 % | Oui |
| Tyrosine | Y | Tyr | UAU, UAC | 3,5 % | 3,3 % | Selon les cas |
| Valine | V | Val | GUU, GUC, GUA, GUG | 6,9 % | 6,8 % | Oui |
| Codon-stop | – | Term | UAA, UAG, UGA | — | — | — |
Fonctions biochimiques
Le tableau ci-dessous propose une brève description des 22 acides aminés classés en fonction des codes et abréviations spécifiés par le comité de nomenclature commun IUPAC - IUBMB :
| Abrév. | Structure | Description sommaire | |
|---|---|---|---|
| A | Ala | 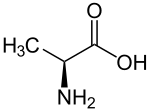 |
Alanine — Abondante et polyvalente. Plus rigide que la glycine, mais assez petite pour ne présenter qu'un encombrement stérique limité pour la conformation de la protéine. Ses résidus peuvent se trouver aussi bien dans le cœur hydrophobe qu'à la surface hydrophile des protéines. |
| B | Asx | Asparagine ou aspartate. | |
| C | Cys |  |
Cystéine — L'atome de soufre de la chaîne latérale se lie facilement aux ions métalliques. En milieu réducteur, deux résidus de cystéine peuvent former un pont disulfure en donnant une molécule de cystine, ce qui stabilise la structure tertiaire. On trouve de tels ponts disulfure dans des protéines agissant dans les environnements extrêmes, comme les enzymes digestives telles que la pepsine et la chymotrypsine, ainsi que des protéines structurelles telles que la kératine. On trouve également de tels ponts disulfure dans les protéines trop petites pour maintenir leur état natif sans leur assistance, comme l'insuline, ou dans les protéines qui ne possèdent pas véritablement de cœur hydrophobe, comme la ribonucléase A. |
| D | Asp |  |
Aspartate — Semblable au glutamate, sa chaîne latérale porte un groupe hydrophile acide fortement chargé négativement. Cet acide aminé est généralement situé sur la surface de la protéine, ce qui contribue à sa solubilité dans l'eau. Ce résidu se lie à des molécules ou à des ions chargés positivement, ce qui est souvent mis à profit par les enzymes pour se lier à des ions métalliques. Lorsqu'il est situé à l'intérieur de la protéine, l'aspartate forme généralement une liaison saline avec un résidu d'arginine ou de lysine. |
| E | Glu |  |
Glutamate — Semblable à l'aspartate, mais avec une chaîne latérale légèrement plus longue et flexible. |
| F | Phe | 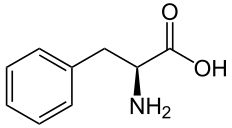 |
Phénylalanine — Essentielle pour l'homme, elle présente un groupe aromatique phényle à l'extrémité de sa chaîne latérale. Il s'agit d'un résidu volumineux et hydrophobe, plutôt présent à l'intérieur des protéines. La phénylalanine peut être convertie en tyrosine par hydroxylation. |
| G | Gly | Glycine — Avec une chaîne latérale réduite à un atome d'hydrogène, c'est le plus simple et le plus petit des acides aminés, et le seul acide aminé protéinogène non chiral. Il ajoute de la flexibilité aux chaînes polypeptidiques. Il peut s'insérer dans les espaces les plus étroits, ce qui permet notamment les structures compactes observées par exemple dans les triples hélices du collagène. | |
| H | His | 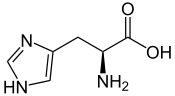 |
Histidine — Essentielle pour l'homme, elle présente un groupe imidazole situé à l'extrémité de sa chaîne latérale et dont l'atome d'azote est protoné même en milieu légèrement acide. Ce résidu intervient dans de nombreuses protéines comme mécanisme régulateur pour en modifier la conformation et le fonctionnement en milieu acide tel qu'observé dans les endosomes et les lysosomes, induisant le changement conformationnel de certaines enzymes. Cet acide aminé demeure cependant relativement rare dans les protéines. |
| I | Ile | 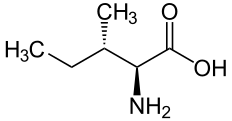 |
Isoleucine — Essentielle pour l'homme, elle présente une chaîne latérale aliphatique. Elle forme des résidus hydrophobes qui jouent un rôle important dans le repliement des protéines et tendent à occuper l'intérieur de leur structure tridimensionnelle. L'isoleucine contient un second carbone asymétrique en position β. Seul l'isomère (2S, 3S) est présent dans les protéines. |
| J | Xle | Leucine ou isoleucine. | |
| K | Lys | 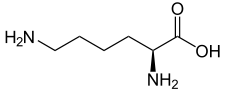 |
Lysine — Essentielle pour l'homme, elle est fonctionnellement semblable à l'arginine. Sa longue chaîne latérale flexible est terminée par une charge électrique positive, ce qui convient à la fixation de molécules chargées négativement. C'est par exemple le cas des protéines qui se lient aux acides nucléiques que sont l'ADN et l'ARN. Les résidus de lysine tendent à se trouver à la surface des protéines ; lorsqu'ils se trouvent à l'intérieur, ils forment généralement des liaisons salines avec des résidus d'aspartate et de glutamate. Il existe de nombreux dérivés de la lysine formés au cours de modifications post-traductionnelles des protéines. |
| L | Leu | 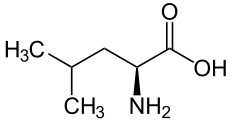 |
Leucine — Essentielle pour l'homme, elle présente une chaîne latérale aliphatique. Elle forme des résidus hydrophobes qui jouent un rôle important dans le repliement des protéines et tendent à occuper l'intérieur de leur structure tridimensionnelle. |
| M | Met |  |
Méthionine — Essentiel pour l'homme, c'est toujours le premier acide aminé incorporé dans les protéines, dont il est parfois retiré lors de modifications post-traductionnelles. Sa chaîne latérale contient un atome de soufre portant un groupe méthyle qui peut être mobilisé dans certaines réactions métaboliques. |
| N | Asn |  |
Asparagine — Semblable à l'aspartate, mais avec un groupe amide –CONH2 à la place du carboxyle –COOH terminal de la chaîne latérale. |
| O | Pyl |  |
Pyrrolysine — Apparentée à la lysine mais avec un groupe azoline à l'extrémité de sa chaîne latérale, elle n'est présente que dans certaines enzymes liées au métabolisme méthanogène de certaines archées. Elle est codée indirectement par le codon-stop UAG Ambre en présence d'un élément PYLIS en aval de ce codon. |
| P | Pro | 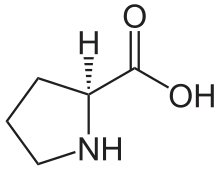 |
Proline — Avec leur amine secondaire intracyclique, les résidus de proline sont rigides et présentent une géométrie différente de celle de tous les autres acides aminés protéinogènes. Pour cette raison, la proline termine généralement les structures secondaires telles que les hélices α et les feuillets β. Elle est abondante dans le collagène sous forme d'hydroxyproline, obtenue par modification post-traductionnelle. |
| Q | Gln | 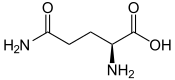 |
Glutamine — Semblable au glutamate, mais avec un groupe amide –CONH2 à la place du carboxyle –COOH terminal de la chaîne latérale. |
| R | Arg |  |
Arginine — Fonctionnellement semblable à la lysine, elle présente une longue chaîne latérale flexible terminée par un groupe guanidine portant une charge électrique positive, ce qui convient à la fixation de molécules chargées négativement. C'est par exemple le cas des protéines qui se lient aux acides nucléiques que sont l'ADN et l'ARN. Les résidus d'arginine tendent à se trouver à la surface des protéines ; lorsqu'ils se trouvent à l'intérieur, ils forment généralement des liaisons salines avec des résidus d'aspartate et de glutamate. |
| S | Ser | 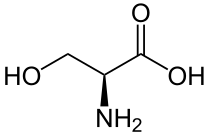 |
Sérine — Fonctionnellement semblable à la thréonine, elle présente une petite chaîne latérale terminée par un groupe hydroxyle –OH, dont l'atome d'hydrogène est labile, de sorte que ce résidu intervient souvent comme donneur d'hydrogène dans les enzymes. La sérine est très hydrophile et se trouve par conséquent préférentiellement à la surface des protéines solubles. |
| T | Thr | 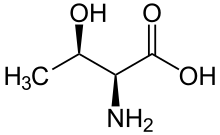 |
Thréonine — Essentiel pour l'homme, cet acide aminé présente une chaîne latérale terminée par un groupe hydroxyle –OH, dont l'atome d'hydrogène est labile, de sorte que ce résidu intervient souvent comme donneur d'hydrogène dans les enzymes. La thréonine est très hydrophile et se trouve par conséquent préférentiellement à la surface des protéines solubles. Elle contient un second carbone asymétrique en position β. Seul l'isomère (2S, 3R) est présent dans les protéines. |
| U | Sec | 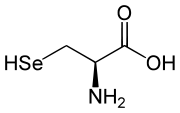 |
Sélénocystéine — Analogue structurel de la cystéine dans lequel un atome de sélénium remplace l'atome de soufre, la sélénocystéine présente un pKa et un potentiel d'oxydoréduction plus faibles que ceux de la cystéine, ce qui convient bien aux protéines antioxydantes[7] |
| V | Val | 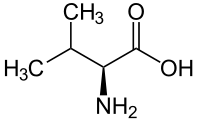 |
Valine — Essentielle pour l'homme, elle présente une chaîne latérale aliphatique. Il forme des résidus hydrophobes qui jouent un rôle important dans le repliement des protéines et tendent à occuper l'intérieur de leur structure tridimensionnelle. |
| W | Trp | 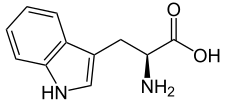 |
Tryptophane — Essentielle pour l'homme, elle présente un groupe aromatique indole à l'extrémité de sa chaîne latérale. Il s'agit d'un résidu volumineux et hydrophobe, plutôt présent à l'intérieur des protéines. Ses résidus présentent une fluorescence solvatochrome. C'est un précurseur de la sérotonine. |
| X | Xaa | Indique la présence d'un acide aminé indéterminé. | |
| Y | Tyr |  |
Tyrosine — Dérivée de la phénylalanine par hydroxylation, la tyrosine présente un groupe aromatique phénol à l'extrémité de sa chaîne latérale. Il s'agit d'un résidu volumineux. C'est un précurseur de la mélanine, de l'adrénaline et des hormones thyroïdiennes. Elle est également fluorescente, bien que cette fluorescence soit généralement interrompue par transfert d'énergie à des résidus de tryptophane. |
| Z | Glx | Glutamate ou glutamine. | |
Origine évolutive
La plupart des bactéries sont capables de synthétiser les vingt acides aminés protéinogènes, mais elles le font suivant deux types de méthodes. Une première série d'acides aminés « primaires » est créée à partir de composés inorganiques. Les acides aminés « secondaires » sont formés à partir des « primaires » qui leur servent d'éléments précurseurs[8].
Les conversions connues entre acides aminés sont les suivantes[9] :
| Précurseur | Composé secondaire |
|---|---|
| Glu | Gln, Pro, Arg |
| Asp | Asn, Thr, Lys |
| Gln | His |
| Thr | Ile, Met |
| Ser | Trp, Cys |
| Val | Leu |
| Phe | Tyr |
Les acides aminés primaires sont moins d'une dizaines. Ce sont eux qui se forment spontanément dans les expériences de chimie prébiotiques, ou que l'on retrouve dans les météorites carbonées[8]. Ces éléments suggèrent qu'ils sont les premiers apparus, et que les premières protéines n'étaient formées que de ceux-ci[8]. Le reste de la série ne relève alors pas de la chimie pré-biotique, mais sont le produit des métabolismes primitifs.
Notes et références
- ↑ (en) Alexandre Ambrogelly, Sotiria Palioura et Dieter Söll, « Natural expansion of the genetic code », Nature Chemical Biology, vol. 3, no 1, , p. 29-35 (PMID 17173027, DOI 10.1038/nchembio847, lire en ligne)
- ↑ (en) Fred Sherman, John W. Stewart et Susumu Tsunasawa, « Methionine or not methionine at the beginning of a protein », BioEssays, vol. 3, no 1, , p. 27-31 (PMID 3024631, DOI 10.1002/bies.950030108, lire en ligne)
- ↑ (en) Francis A. Carey, « Table of pKa and pI values », sur Département de chimie de l'université de Calgary, (consulté le )
- ↑ La cystéine est une molécule polaire, cependant les résidus de cystéine des protéines sont souvent impliqués dans des interactions hydrophobes et assurent de ce fait une fonction biochimique essentiellement apolaire :
(en) P. Heitmann, « A Model for Sulfhydryl Groups in Proteins. Hydrophobic Interactions of the Cysteine Side Chain in Micelles », European Journal of Biochemistry, vol. 3, no 3, , p. 346-350 (PMID 5650851, DOI 10.1111/j.1432-1033.1968.tb19535.x, lire en ligne) - ↑ (en) Paula Yurkanis Bruice, Organic Chemistry, Pearson Education Inc., , 4e éd. (ISBN 0-13-121730-5), p. 960-962
- ↑ (en) M. Beals, L. Gross, S. Harrell, « Amino Acid Frequency », sur The Institute for Environmental Modeling (TIEM) à l'université du Tennessee (consulté le )
- ↑ (en) Byung Jin Byun et Young Kee Kang, « Conformational preferences and pKa value of selenocysteine residue », Biopolymers, vol. 95, no 5, , p. 345-353 (PMID 21213257, DOI 10.1002/bip.21581, lire en ligne)
- 1 2 3 Code Biology – A New Science of Life. Marcello Barbieri Biosemiotics 5(3) · December 2012.
- ↑ ACo-EvolutionTheoryoftheGeneticCode. Wong, J. T. F. Proc.Nat.Acad.Sci.USA Vol.72, No.5, pp.1909-1912, May1975.
Liens externes
- Gilles Camus, « Les acides aminés », sur UFR de Biologie de l'université Pierre-et-Marie-Curie, (consulté le )
- L'origine du code à une lettre pour les acides aminés

.png.webp)